「グローバル」な発想で : Mascot Distiller 2.8 におけるラベルフリー定量法
Mascot Distiller(定量モジュール)ではプリカーサーベースの定量計算が可能です。DDA、precursorデータによるラベルフリーの手法としては、弊社にて「Replicate」と「Average」と呼んでいる2種類の方法をサポートしています。version 2.8では、このうちReplicate法について様々な改良を行いました。これは、質量と溶出時間でアライメントされた複数のデータセットに含まれるプリカーサーのeXtracted Ion Chromatograms(XICs)の情報を利用した定量解析方法です。
Mascot 2.7以前のreplicate 定量
Mascot Distiller 2.8 より前のバージョンでは、'Replicate' 定量データセットの処理を行う場合、データ読み込み時に'memory efficient'という名称のチェックボックスをオフにして、単一のプロジェクトとして計算を実行する必要がありました。計算対象のrawデータを1つのプロジェクトファイル(解析単位のdistillerファイル)にてまとめて読み込みます。各rawデータから抽出したピークリストを1つにまとめ上げたmgfファイルを作成してMascotで検索し結果ファイルを得ます。この時同定されたすべてのペプチドに対するXIC検出を,すべてのrawデータに対して行います。同定ペプチドが必ずしもすべてのrawデータで直接的に含まれているとは限りません。Rawデータ内に同定ペプチドが含まれない場合、他のファイルで同定されたXICピークの位置(保持時間)を出発点として、ユーザーが定義した設定値の間にシフトしたXICピークとみなせるものがないか、前後の時間帯から該当プリカーサーを探します(設定値デフォルトは500秒)。この処理で識別された最も強い箇所を比較ポイントとみなし、XICにてアラインメントされ、定量値並びにペプチド比を計算していきます。
Mascot Distiller 2.8でのreplicate 定量
Mascot Distiller 2.8では、XIC検出の前に「グローバルタイムアライメント」計算が実施されます。アライメント結果は、ペプチドが直接同定できなかったrawデータにおいて、該当ペプチドのXIC検出の開始点として使用されます。この仕組みによりそれぞれのrawデータで独立した計算を実行する事ができるようになりました。
アライメントはまず各rawデータをラフに並べ、トータルイオンクロマトグラム(TIC)の情報なども用いながら”consensus dataset”を作成するところから始めます。Consensus datasetの情報を足掛かりに、信号処理への適用を目的とした改良型の再帰的相互相関法(recursive cross correlation method)を適用して候補のペアを評価しながら最初のアライメントを実行します。この計算を経てconsensus datasetからさらに確度が高いコンセンサス(features)が特定され、詳細なアライメントの際の起点とされます。新たなアライメントは、再帰的相互相関法と最小二乗法を用いた最適化アルゴリズムの組み合わせを使用して実行されます。すべてのコンセンサス(features)情報を使って一対のプロジェクト間のタイムシフトを計算します。
この方法の利点は以下の通りです。
- 個々のサンプルを個別に処理・検索する事ができ、(これまでのようにすべてのrawファイルを同時に開かなくて済むため)メモリ使用量が少なくて済みます。XICの検出も個別に行われるため、より効率的な並列化が可能になります。
- 計算されたタイムアライメントシフトが使用されるため、ユーザー側でデータセットのタイムシフトに関するパラメータを入力する必要がなくなり、結果がより客観的なものとなります。
同等のハードウェアで同じサンプルを処理した我々のテストでは,主にXIC検出の並列化がもたらした改善効果で,Distiller 2.8はDistiller 2.7.1に比べて 2~3倍速くなり,メモリの最大使用量も約1/3になりました。
解析例
Distiller 2.8でReplicate手法の定量性をテストするために、 PRIDEプロジェクトPXD001385 から12個のrawファイルをダウンロードしました。Shalitら1は、このデータセットを使って、ExpressionistとMaxQuantという2つのLFQ(Label Free Quantitation)ソフトウェアで計算されたサンプル量比と強度を比較しました。プロジェクトの説明文によりますと、3、7.5、10、15と呼ばれる4つのグループそれぞれについて3つのreplicates(繰り返し実験・測定結果)データが準備されています (訳者注:4種類 x 3回繰り返しという事で合計12のrawデータが公開されています)。各数字は、200ngのHeLa消化物にスパイクされた大腸菌の量(ナノグラム単位)を示しています。これにより、15ngのサンプルとの量比が 5(3ng)、2(7.5ng)、1.5(10ng)となる事が期待されたデータです。
ファイルはMascot Distiller 2.8上で処理をし、検索は Mascot Server 2.7で行っています。Distillerの処理においては(データ測定装置である)Thermo Q Exactiveデータ用のパラメータを使用しています。検索対象のデータベースはヒトおよび大腸菌のプロテオームデータ並びにコンタミントデータベースを合わせたものを使用しました。1%のペプチドFDRを同定基準としたところ、2072個のヒトタンパク質と317個の大腸菌タンパク質が同定されました。定量計算としては15ngサンプルと各サンプルを比較した際、大腸菌由来のタンパク質では5:1(3ng)、2:1(7.5ng)、1.5:1(10ng)に、ヒト由来のタンパク質は1:1になるか、という観点から結果を評価しています。参照論文と同様、ペプチド比は、20のヒトタンパク質情報を使って正規化しました。MASCOT ServerないしはDistillerのオプションで正規化の対象となるタンパク質の指定が可能で、選別されたタンパク質はintensityが全体の中でちょうど真ん中あたりの強度のものを選んでいます。参照論文では、様々な方法でタンパク質の定量値並びに比を計算しています。Mascot Distillerでのタンパク質比の計算は、アサインペプチド比の平均値または中央値にて算出出来るほか、今回の計算でも採用した「Weighted」という選択肢もあります。「Weigthed」は各ペプチドのintensity情報を重みづけに利用した計算です。ただし今回のデータについては、後の検証により中央値を用いた結果とあまり差がなかった事を確認しています。
大腸菌のタンパク質比率について、主成分分析(PCA)を用いて複製間の定量再現性の可視化を試みました。計算並びに図の作成にはMascot Distiller 2.8に追加された新しいレポート機能を利用しています(Figure 1)。「Target ratio 2」グループ(15ng:7.5ng)の実験データ1つに異常値がある以外は、各グループ内でsampleデータがまとまっており、高い再現性を示唆しています(訳者注:グラフのプロットの同じ色が同じ大腸菌注入量で、グラフの近い場所にあるデータは各タンパク質のratioの組み合わせも似ていることを示します。同じ色(大腸菌のロード量が同じサンプル)のプロットが似たような場所にあるので、結果の再現性が高いことが示唆されています)。これは、Shalitら1が行った同様のPCA分析と一致しています。そちらの解析でも7.5ngのサンプルの1つが異常値として特定されています。
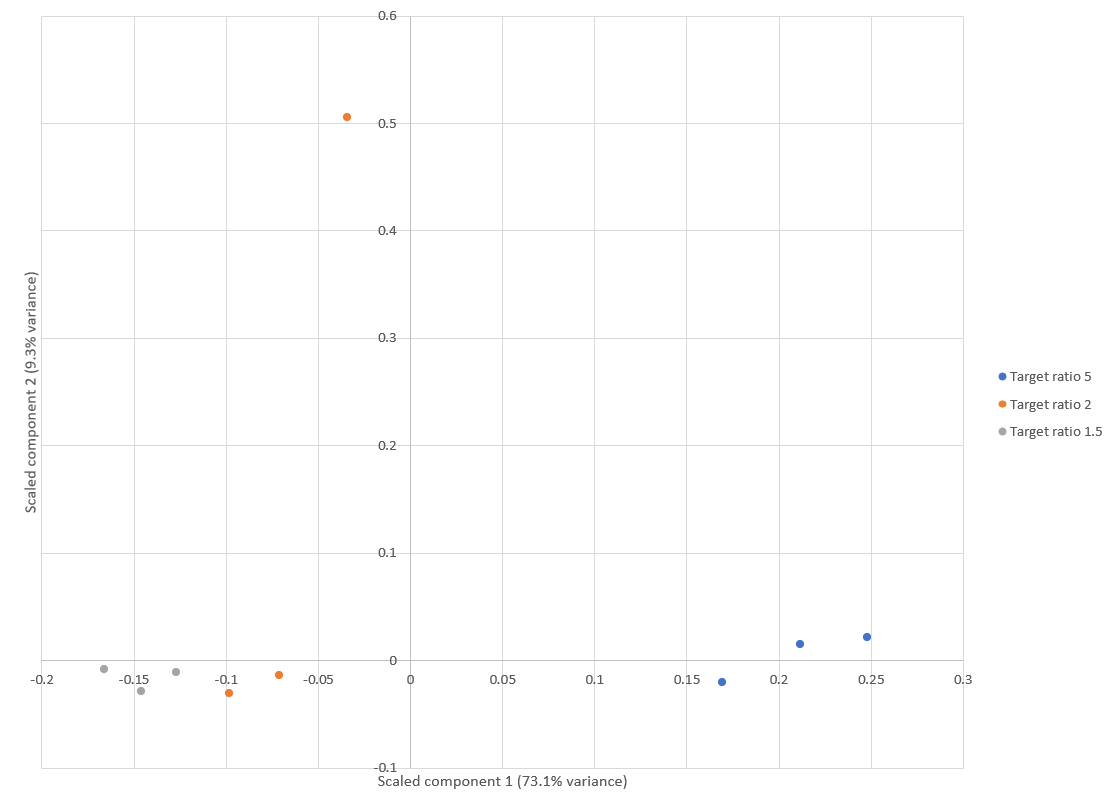
Click to view full size image
Figure.1:大腸菌のタンパク質比率を用いたPCAによるスコアプロット。「Target ratio 2」のグループの1データを除き、すべての各サンプルが他のグループから離れた場所で集まっています。これはShalitら1の参照論文の figure 3と同じ計算を行った図です。
定量データの精度を調べるために、大腸菌で同定されたタンパク質のうち、2つ以上の定量値をもつペプチドがアサインされたタンパク質のratioの中央値を、各サンプル(ratio:5、2、1.5)において計算しました。その結果を以下のFigure 2に示します。
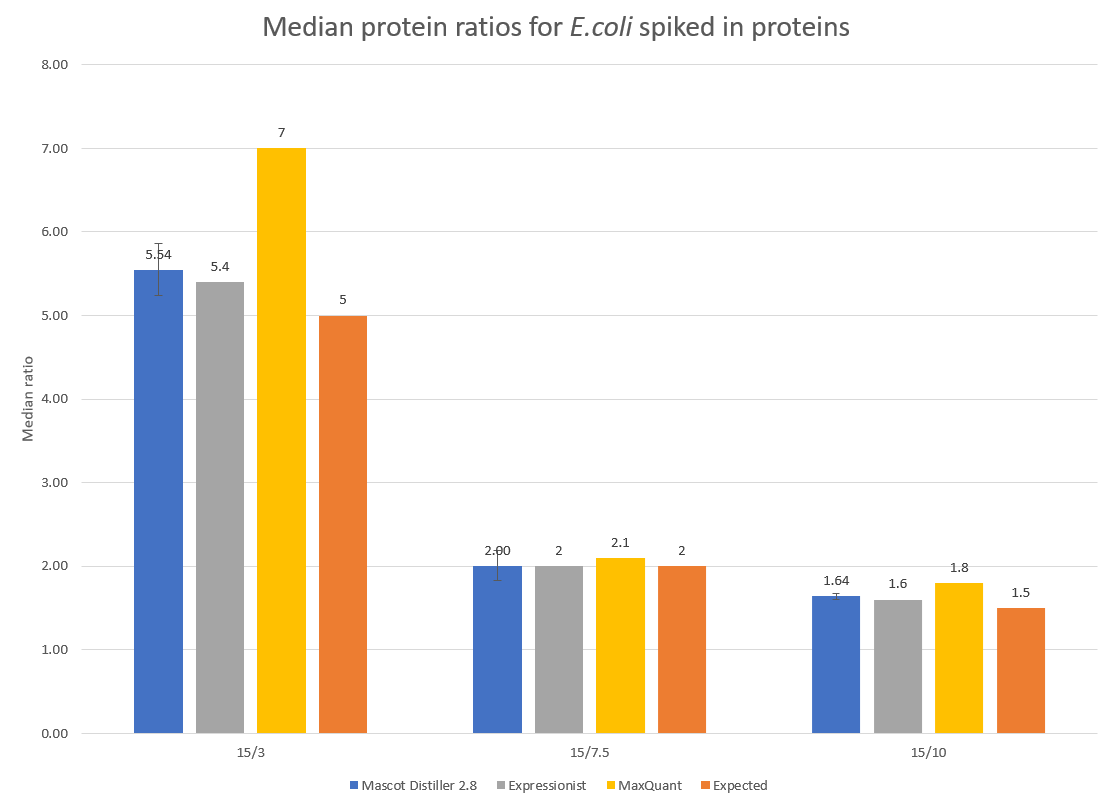
Click to view full size image
Figure 2: 大腸菌に混入したタンパク質の比について、検出値と期待値の比較。エラーバーは95%信頼区間を示しています。ExpressionistとMaxQuantの中央値が比較のために示されていますが、これはShalitら1の論文にあるfigure 8dをもとに作成され、図内で表記されている数字を引用しています。
このようにMascot Distiller 2.8では旧バージョンのソフトウェアと比較して、速度とメモリ使用量の改善が行われ、ラベルフリー定量において良好な精度と再現性が得られている事が確認できました。
すでにMascot Distillerのライセンスをお持ちの方は、2.8に無料でアップデートできます(訳者注:Distillerは機能別にモジュール構成となっています。定量計算を行うためにはDistiller定量モジュールをご購入いただいている必要があります)。お持ちでない方は30日間の試用版をご用意しておりますのでぜひお試しください。詳細は弊社までご連絡ください。
Reference
[訳者補足1]
Referenceの論文では、MaxQuantとGeneData Expressionist の比較を行っておりますが、その際に定量データを評価するいくつかのポイントについて説明されており、ご自身が定量解析を行う上で参考になると思います。また、iBAQやHi-Nといった、タンパク質にアサインされたプリカーサーのピーク強度を比較的簡単に利用した定量指標/数値 についても併せて評価しており、その結果から簡易的なラベルフリー定量でも実用的なレベルで使用できる事を主張しています。
[訳者補足2]
PXD001385 のデータを使って、今回のブログ記事またはReferenceの内容を参考に作成した日本語チュートリアル資料を作成しました。
MASCOT Distiller Replicateラベルフリー定量解析チュートリアル
Keywords: global time-alignment, label-free, Mascot Distiller, quantitation, replicate, retention time