イオンモビリティフィルタリングによるプリカーサー定量結果の改善
Mascot Distiller 2.8の主な新機能の1つとして、Bruker社 timsTOFからのrawデータ読み込みに対するサポートが挙げられます。timsTOFはイオンモビリティとQTOFを組み合わせて、分離に関して新たな次元の追加を可能にします。 isobaricペプチドのスペクトル分離にも利用できます。
SILAC や Label Free などのプリカーサーベースの定量法の XIC ピーク検出時、Mascot Distiller 2.8 では同定されたペプチドのプリカーサーイオンモビリティ値を使用したプリカーサーの分離をする事ができます。対象となるプリカーサー領域のデータについて干渉信号を除去して精度を高め、より信頼性の高い定量結果を得る事が分離の目的です。計算に使用するよう許容されたイオンモビリティ範囲内のMS1シグナルのみが、XICのピーク検出に使用されます。
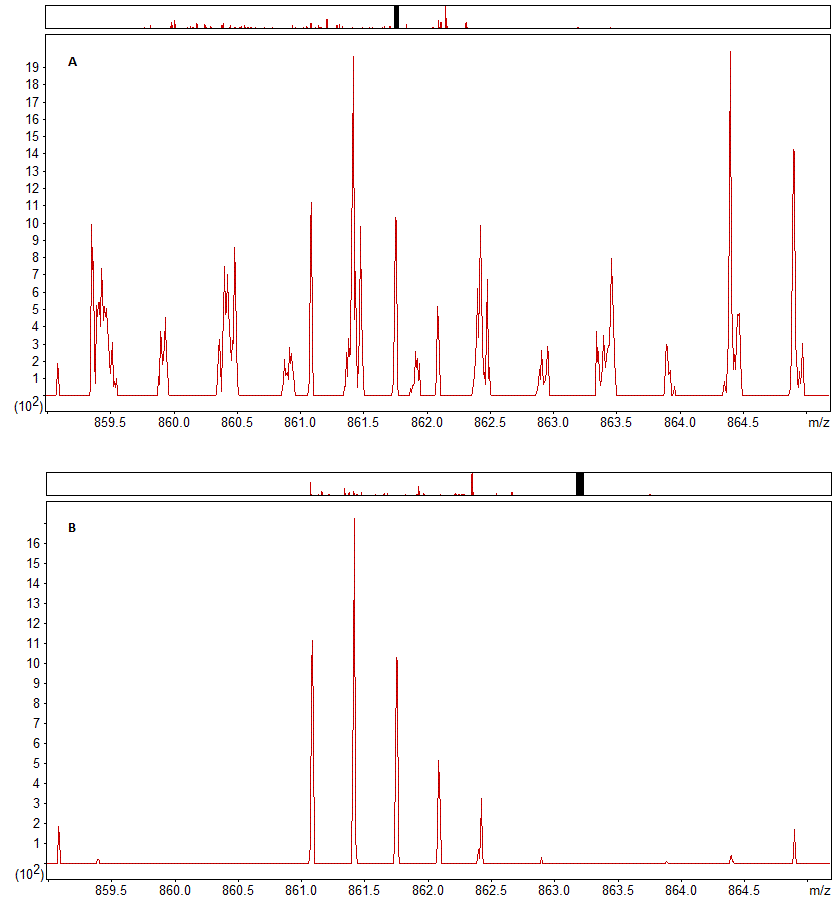
このプリカーサーイオンモビリティフィルタリングの効果を示しているのが下の図1です。フィルタリングを行った場合と行わなかった場合のサンプルの1回のスキャンから得られたペプチドのプリカーサー領域を示しています。 ご覧のようにイオンモビリティフィルタリングを有効にすると、非常に強度の低いプリカーサーであるにもかかわらず、プリカーサー領域の信号ははるかにクリーンでノイズピークも少ないことがわかります。
Click to view full size image
図1:プリカーサーイオンモビリティフィルタリング(A)を無効にした場合と(B)を有効にした場合の、強度が低い 3+ペプチド領域の様子。
フィルタリングの効果は、fractionとcorrelationという、Distillerで採用されているXICs計算に関連するMS1ピークマッチングの評価に関するパラメータにも表れています。Fractionはプリカーサー領域の全ピーク面積のうち、予想されるプリカーサーピークが占める割合です。またcorrelationは、ペプチドのプリカーサー同位体分布の予測値と観測値の重ね合わせを検証した際の相関係数となります。図1で示した領域はイオンモビリティのフィルタリング適用により、fractionが0.2978から0.9234に、correlationが0.6723から0.9463に改善しています。
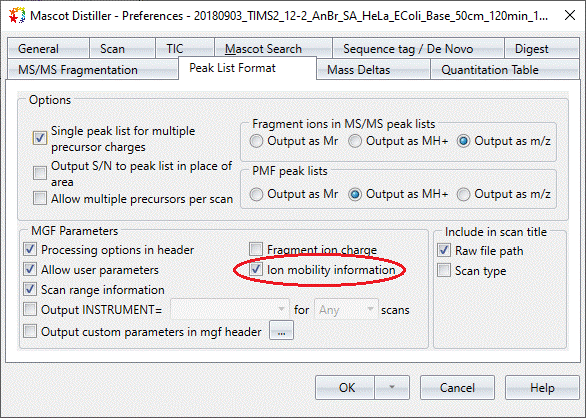
実際にフィルタリングを行うためにはMASCOT Serverのバージョンが2.7以上になっている必要があります。MGFにイオンモビリティ値を出力する必要がありますが、図 2 に示すように、Distillerの"Preferences" ダイアログの "Peak List Format" タブにあるオプションにチェックを入れてください。
Click to view full size image
図2:Mascot Distiller 2.8のPeak List Formatオプションで、ピークリストのプリカーサーイオンモビリティ値をエクスポートするオプションの位置に赤い丸を付けています。
Mascot DistillerをMascot Daemonのdata import filterとして使用する場合、以下の手順でイオンモビリティ機能を有効にする必要があります。
- WindowsにAdministrator権限を持つユーザーとしてログイン
- Windowsの検索バーに「cmd」と入力
- 「Command Prompt」の上で右クリックし、「管理者として実行」をクリック
- CdコマンドなどでMascotDistiller.exeが格納されているフォルダに移動(デフォルトではC:Program Files\Matrix Science\Mascot Distillerです)
- 以下のコマンドを実行
MascotDistiller.exe -batch -daemonMGFIonMobilityParam 1
[訳者注:近日予定しているMASCOT Server/Daemon のアップグレードによってより簡単に対応できるようになる予定です]
大腸菌スパイクしたサンプルの例
プリカーサーイオンのモビリティフィルタリングの効果を調べるために、PXD010012から2つの複製を示す4つのファイルを取得しました1。 これらのサンプルは、HeLaタンパク質ダイジェストをバックグラウンドにして、大腸菌タンパク質ダイジェストをスパイクしたものです。 著者らは、タンパク質に混入した大腸菌の比率のmedianを4.31と報告しています。
選択されたrawファイルは、Mascot Distillerを用いてTimsTOFデータ向けのピーク抽出オプション(optファイル)で処理され、2回検索されました 。ピークリストにプリカーサーイオンモビリティ値が含まれる場合と含まれない場合の結果を比較します。
各検索で同定されたすべてのタンパク質は、Mascot Distiller を使用して定量化され、最も強く同定された 20 種類のヒトタンパク質とのペプチドマッチを使用して、ペプチド比を正規化しました。 これにより、定量化の際にプリカーサーイオンモビリティフィルタリングを行った場合と行わなかった場合の2つの結果が得られました。 結果は以下の表1にまとめられています。
| Ion mobility filtering disabled | Ion mobility filtering enabled | |
|---|---|---|
| 検索時間(hours) | 36 | 10 |
| human proteinのratio : mediana,b | 0.979 | 1.017 |
| 定量計算されたhuman protein の数a | 4458 | 4946 |
| E.coli proteinのratio : mediana,c | 4.21 | 4.30 |
| 定量計算されたE.coli proteinの数a | 718 | 821 |
| fraction : median | 0.487 | 0.686 |
| correlation : median | 0.912 | 0.959 |
| 各タンパク質の定量計算に使用されたペプチド数 : median | 5 | 6 |
a 2つ以上の異なるペプチドが有効な比を持つタンパク質比のみがカウントされています。 b 比の値が1.0になる事が想定されたデータです1。 c 論文でレポートされたratioは4.31です。
このようにイオンモビリティによるフィルタリングを実行すると結果が改善されます。またフィルタリングを有効にする事で定量計算の速度も大幅に速くなりました。これは、イオンモビリティのスライスを個別に取得することで、処理しなければならないデータが少なくなったためです。 ペプチドのfraction、correlationについてmedianを比較しても改善された事がわかります。これはフィルタリングを有効にすることで、データの品質とS/Nが向上し、より多くのペプチドratioが定量計算を行うための条件としている閾値を通過できた質の良いデータが増えたことを示しています。 タンパク質ratioの算出に使用したペプチド配列の数(median)も5から6に増加し、少なくとも存在確度の高いペプチド配列を持つタンパク質がより多く定量されました。
すでにMascot Distillerのライセンスをお持ちの方は、2.8を無料でアップデートできます。お持ちでない方は、Mascot Distillerの30日間のトライアルをご利用いただけます。詳細は、弊社までご連絡ください。
Reference
Keywords: ion-mobility, Mascot Distiller, quantitation, timsTOF