SARS-Cov-2 スパイクタンパク質のジスルフィド結合の構造解析
SARS-Cov-2ウイルスに対して、2年前に発見されて以来数多くの研究が行われています。現在適用可能なほぼすべての分析方法が使用されており、PubMedによると3000を超えるプロテオミクスの論文が掲載されているようです。免疫学的に最も重要なSARS-Cov-2のタンパク質は、標的細胞への最初の結合に関与するスパイクタンパク質です。包括的なスパイクタンパク質の解析例として、論文「Virus-Receptor Interactions of Glycosylated SARSCoV-2 Spike and Human ACE2 Receptor」があります。この研究では、タンパク質の配列、グリコシル化、ジスルフィド結合といった内容について調べ、それらを3Dモデリングと相互作用の研究に適用しています。この論文の解析データはPRIDEプロジェクトPXD019939で公開されています。このブログ記事は、Mascot Server 2.7で導入されたクロスリンク結合検索機能を用いて論文にあるジスルフィド結合のデータを再解析したものです。
解析に使用したスパイクタンパク質は、免疫反応を刺激する事が可能な模倣免疫原として設計されたプロトタイプのDNAワクチンから精製したものです(参考文献)。精製タンパク質は還元せず、アルキル化した後、トリプシン、Lys-C、Glu-Cを組み合わせた消化酵素処理(EKRZ)、またはα-Lytic Proteaseによる酵素消化を行いました。またMascot Server上でこれら設定に合う切断パターンのEnzyme設定を作成しました。紹介論文では消化処理後のスパイクタンパク質上のグリコシル化構造に関する詳細な研究も行っていますが、ジスルフィド結合解析の邪魔にならないよう、タンパク質はPNGaseFを用いて脱グリコシル化されています。
測定はThermo Orbitrap Fusion Lumosを使い、ETDフラグメンテーションで行われました。2つのデータセットはそれぞれSARS-CoV-2、contaminants、Uniprot Humanのデータベースに対して検索しましたが、これらのデータベースはMascotのpredefined データベースとして設定などが予め準備されており、すぐに利用可能です。測定のrawデータを、Mascot DistillerでThermoFisher社製質量分析装置データのデフォルトオプションを使って処理しました。クロスリンクペプチドの多くは、一般的なペプチド解析時よりも大きい質量と電荷状態を持っているので、フラグメントイオンをデチャージし、一価のピーク位置に換算した値を生成するように設定変更しました。これにより、3+以上のフラグメントイオンが存在する場合でもそれらのピークを理論値とのマッチングに利用することができます。
この論文で著者らは、タンパク質全長の中でペプチドが同定された箇所の特定とクロスリンク検索を組み合わせる事で、シグナルペプチドが切断された位置が13番目グルタミンである事を特定しました。その解析結果を踏まえ私たちはこのスパイクタンパク質の配列からシグナルペプチドを除いたカスタムデータベースを作成し検索を起こったところ、使用する酵素切断条件下で切断されたN末端が同定ペプチドとして認識される事を確認しました(訳者注:すなわち解析データがシグナルペプチド切断箇所の情報について正しく反映している事を確認しました)。解析に利用したMASCOTのcrosslinking設定は、デフォルトで準備されている3設定のうちの1つ「Disulfide bridge in Lysozyme」を出発点とし、検索対象のタンパク質としてスパイクタンパク質の配列「SPIKE_SARS」とそのシグナルペプチドを除いたオリジナルのエントリーを検索対象とするようアクセッション番号を設定しました。
検索条件は以下の通りです。
Crosslinking : Disulfide bridge in SARS2 Spike glycoprotein Enzyme : Trypsin+Lys-C+Glu-C Variable modifications : Carbamidomethyl (C), Oxidation (M), Gln->pyro-Glu (N-term Q), Deamidated (NQ) Linkers : Xlink:Disulfide (C) Peptide mass tolerance : ± 10 ppm Fragment mass tolerance: ± 0.1 Da Max missed cleavages : 2 Instrument type : ETD-TRAP
酵素処理2種類のデータを検索した結果、論文を読んだ際に予想していたよりもはるかに多くのジスルフィド結合を検出する事ができました。 解析後、同定タンパク質をFASTA形式でまとめたファイル、MGFファイル、xiVIEW csv形式の3種類のファイルを出力しました(以下図参照)。
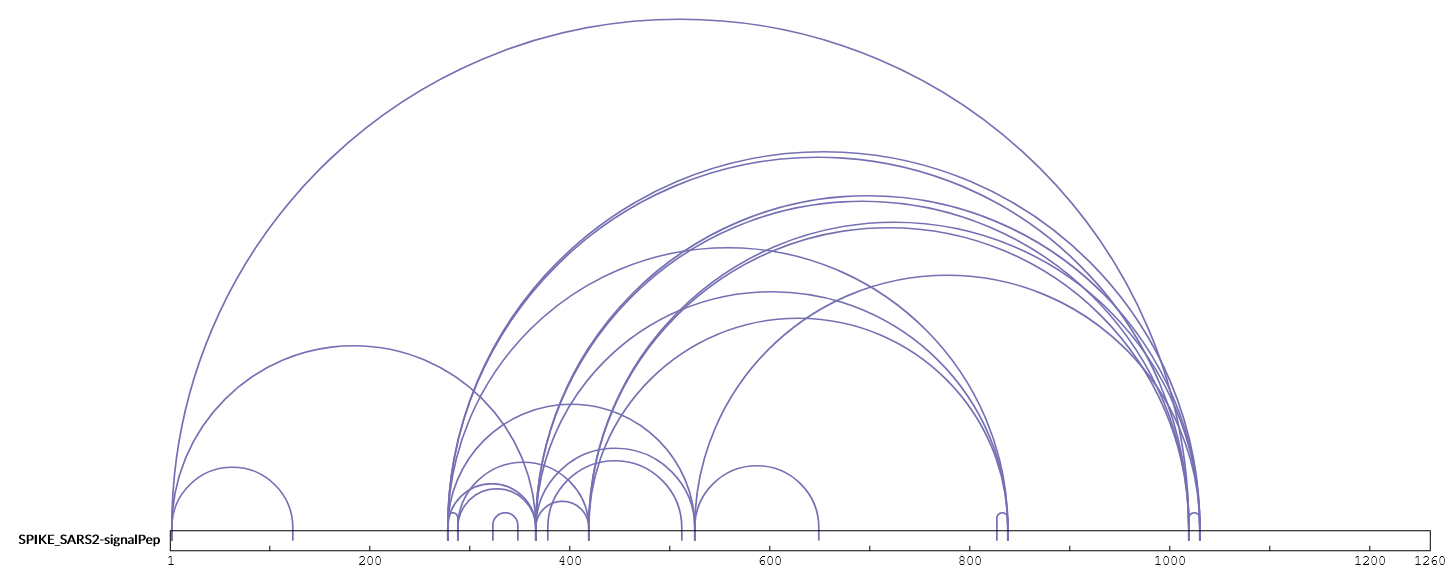
続いて、xiVIEWで新しいデータセットを作成し、先ほどMASCOTから出力したMGF、CSV、FASTAの3ファイルをアップロードして、結果を可視化しました(下図)。
著者らは論文でCys15とCys136の間のジスルフィド結合のみ報告しています。これは、シグナルペプチドの切断がCys15の前に起こったことを証明する上で重要なものですが、今回私たちが行った検索結果からは他のジスルフィド結合の可能性が同定されていることが分かります。Mascot Serverで得られた結果を、Uniprotに記録されているスパイクタンパク質の既知のジスルフィド結合と比較したところ、Mascot Serverは28のジスルフィド結合を同定し、そのうち8つがUniProtの既知情報と一致しました。分析した試料はSARS-Cov-2ウイルス調製物から抽出・精製した材料ではなく模倣免疫原であったため、残り20のジスルフィド結合が自然に発生し得るかどうか、はっきりしたことは言えません。しかし模倣免疫原を使用するコンセプトからは、少なくとも自然界に存在してもおかしくないと言えます。
それでは具体的に、MASCOTで同定されUniProtに掲載されていなかった高スコアのマッチング内容をいくつか見ていきましょう。LPDDFTGCVIAWNSNNLDSK C8<-Xlink:Disulfide->C1 CTLK, アミノ酸位置301-432のジスルフィド結合という結果は、Mascotスコアが78で期待値5.8e-8と良好な一致と言えます。ジスルフィド結合の両ペプチドで、N末端→C末端もC末端→N末端のどちらの向きのフラグメントも、十分にマッチしている事がわかります。
我々は以前ブログで、インタクトな架橋ペプチドのマッチングを検証するためのガイドラインとなる記事(英語、日本語)を公開しており、今回もその方針でマッチングを評価しました。上記のマッチは、電荷状態が2+と3+で同定されています。どちらの場合もランク2位のマッチは1位のマッチと比べると明確に低い低いスコアであり、2位以下の結果は有意なスコアではありません。1位のマッチング内容はかなり確からしいと言えます。
また以下の図はもう一つの例、YNENGTITDAVDCALDPLSETK.C C13<-Xlink:Disulfide->C1 K.CYGVSPTK.L + Deamidated, アミノ酸位置291-379のジスルフィド結合の結果です。Mascot score 72、期待値 1.7e-7 でした。
この結果は、電荷状態4+の1種類のみが同定されました。ランク2位の結果は1位と比べスコア差が10以下、そこから3位の結果はスコア差が2です。ランク2とランク3の配列情報を見ると、ジスルフィド結合の位置は同じであるものの、脱アミノ化の位置が変わり、N4がN2よりわずかに好ましいと判断されているようです。微妙な差があるもののジスルフィド結合については十分な信頼性があると言えます。
SARS-CoV-2 Spikeタンパク質に関するデータベースが時間とともにどのように拡張されていくか、そして今回のように、今後の解析で新たに示唆されたジスルフィド結合が報告されていくのかどうか、興味深いところです。
Keywords: crosslink, SARS-Cov-2