検索エンジンが示す同定の根拠
タンパク質/ペプチド同定のための検索結果の検証とは、以下のように言い換える事ができます。
- タンパク質同定の根拠は何か?
- 同定タンパク質となった根拠となるペプチドは何か?
- 同定を行ったアルゴリズムは、どのような選択肢を検討したのか?
- 同定としたアルゴリズムの判断は正しいか?
これらは、ソフトウェア主導のアプローチによるMS/MSデータ解析において基本的で重要な疑問といえます。MASCOT関連製品では、検索結果をいつでも掘り下げ同定結果の中身を精査していく事が可能です。
ペプチドの同定はボトムアップ型LC-MS/MSの基本です。MASCOT Serverはペプチドを同定する際、何か1つのMS/MSフラグメントピークの存在にこだわって解析することはありません。マッチングにおいてプログラムはまず入力データ選定のため、スペクトルデータを100Daの分割ウィンドウ(ビン)に分け、各分割ウィンドウ内の中で最も高いピークを選択します。選ばれたピークは各ペプチド配列から生成された理論フラグメントピークパターンとマッチングを行い、確率論ベースのアルゴリズムで評価されたスコアが算出されます。続いて、入力データについて分割ウィンドウ(ビン)単位で既に選ばれたピークの次に強度が大きいピークを含んだ新たなピークリストが作成され理論値とのマッチングとスコアリングを繰り返していき、スコアの向上が止まるまで繰り返されます。この方法はMS/MSスペクトル全体で顕著なピークを中心としたアプローチであり、フラグメントピークの欠損やノイズに強い方法といえます。その一方必ずしもすべての理論フラグメントにマッチしない事もあり、ピークが存在していてもそれが入力データとして選定されていなければ、理論値とのマッチングに使われない事もあります。
Click to view full size image
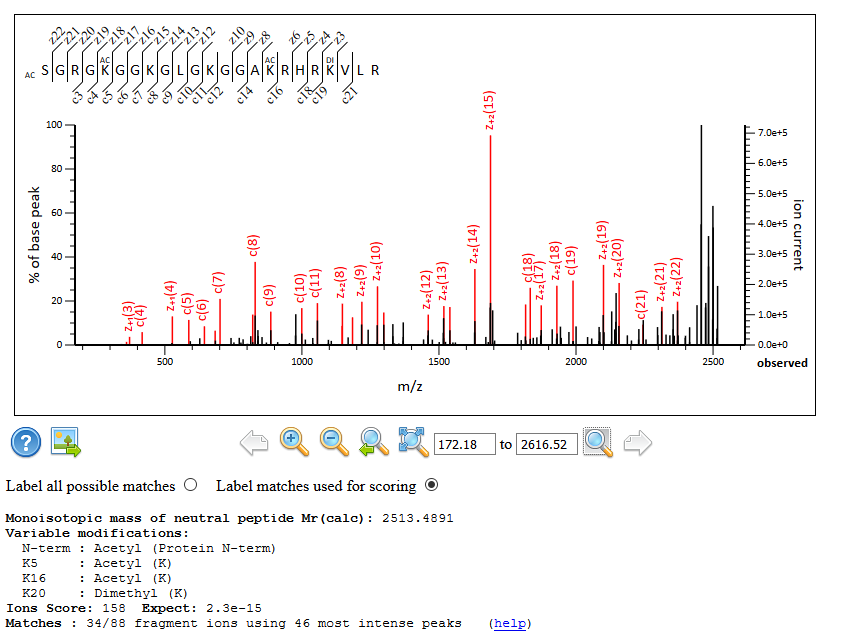
図1:Peptide View画面で表示されるSpectrum Viewerのスクリーンショット。過去のブログ記事「Mascot 2.7のVariable Modifications」(日本語・英語)から。
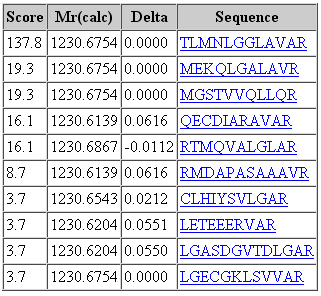
実測値と理論値のマッチング内容はMASCOTのPeptide View画面で確認できます。MASCOT では各スペクトルのスコア上位10件についてその内容を記録しており、Peptide View画面から辿る事で比較する事ができます。1位のペプチドのマッチングについてその内容をすべて手作業で検証するのはさすがに現実的ではありませんが、いくつかをピックアップして確認する事をお勧めいたします。そうする事でシステムに何らかの問題が生じていたり、ピーク抽出に問題が起きていたりする事を発見できる可能性があるからです。また場合によっては2~10位 のマッチングを見る事で、配列や修飾位置の曖昧さを見つけたり、検索パラメータやデータベースの選択に問題があった際に発見できたりする可能性があります。
Click to view full size image
図2:スペクトルマッチングした配列のスコア上位10個のスクリーンショット。過去のブログ記事「High-mass accuracy: fragments」より
個々のデータについてのチェック・結果の検証を行った後、ペプチドマッチに関して検索全体レベルでみた統計情報も併せて確認をしてください。エラー率を使って同定基準の調整を行うために、Target-Decoy検索の実行とFDRの算出をお勧めしています。 この検索を実施するとMASCOTはその場でTargetデータベース(検索で使用した通常のデータベース)のエントリー内容に対応したDecoyデータベース(Target配列をランダム又は逆向きにしたデータベース)を生成し、両者で検索を行います。Decoyデータベースの検索結果はランダムマッチのスコア分布として利用します。

Click to view full size image
図3:MASCOTで利用可能な FDR計算における設定項目について。過去のブログ記事Back to basics 5: Peptide-spectrum match statisticsより。
Decoyデータベースで検索した結果のペプチドやタンパク質を直接見て確認する事も出来ます。結果画面Protein Family Summaryにおいて、「Sensitivity and FDR」のセクションをクリックして展開し、「the decoy report」リンクをクリックする事で、Decoyデータベースにおける検索結果を通常の結果と同様にまとめられた内容で確認できます。TargetとDecoyのマッチング内容を比較する事で、FDR(False Discovery Rate)によって引き直された同定基準値が妥当な値かを確認するのに役立ちます。(訳者注:特にDecoy結果のスコア分布がいつもに比べ高いまたは低い方に偏っている場合、同定基準のラインが極端に変わり同定数の違いに繋がっていることがあります)
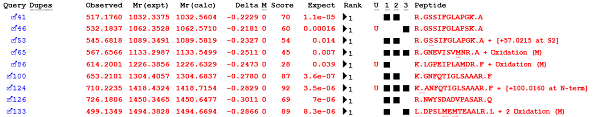
続いてタンパク質同定についてです。基本的な問題として、同定タンパク質として認定される条件が、そのタンパク質にアサインされているペプチドの情報とどのように結びついているのかが1つのポイントです。MASCOTでは同定基準を超えるペプチドのみをタンパク質同定の根拠として使用します。同定ペプチドの基準は一般的にFDRが特定の値(1%など)となるように調整されています。同定タンパク質はさらにシェアペプチドをもつタンパク質をファミリーとしてグループ化します。ファミリーの中に含まれるタンパク質は少なくともそれぞれが1つのユニークな同定ペプチドを持っていて、それがサンプルに含まれる可能性が高いと言える根拠となっています。
Click to view full size image
図4:アルカリホスファターゼのシェアペプチドとユニークペプチドの内容を表す画面のスクリーンショット。Error tolerant 検索の検索結果例画面から
MASCOTのデフォルト設定ではユニークペプチドが最低1つアサインされたタンパク質を同定タンパク質として認定し表示します。最低限必要なアサインペプチド数については、”Format control”を使って2やそれ以上の数に変更可能です。またprotein FDR を計算する時は、グループ(ファミリー)をまとめて1つとして、グループ単位でカウントをします。最低限のアサインペプチド数やprotein FDRといった基準に満たなかったタンパク質にアサインされていたペプチドは、たとえ高いスコアであっても”unassigned”ペプチドリストに移動する事になります。
ユニークペプチドを持たないタンパク質というのは、他のタンパク質でなくそのタンパク質が確かに存在するかどうかの判断が困難です。 Protein Family Summaryでは、そういったsamesetとsubsetのタンパク質も表示させる事ができます。samesetタンパク質とは複数タンパク質で全く同じペプチドマッチの内容になっているタンパク質の事をさします。ペプチドレベルで確たる存在の証拠がないため、どちらか一方、または両方がサンプルに含まれている可能性がありますがそのいずれであるかを特定する事ができません。samesetの場合MASCOT はそのいずれかを代表タンパク質として表示しますが、優先して表示させる生物種を何にするかについては設定が可能です。一方subsetタンパク質とはペプチドのマッチングの一部が同じで(包含関係的に一方に含まれるおり)、かつユニークなペプチドを持たないタンパク質の事です。samesetまたはsubsetタンパク質に含まれるもののいずれかの同定について確度を高めるためには、同定基準を超えるユニークペプチド(特定のタンパク質にのみアサインされる同定ペプチド)の存在が必要です。特定が必要な場合、セットの中に含まれるタンパク質の配列の内容を比較しながら、実験で行う消化酵素処理の酵素の種類を変えユニークペプチドを生み出す事を検討する事で解決する可能性があります。無論単にそのような処理がされるだけでは駄目で、そのペプチドがMS/MSで検出されさらに検索エンジンが提示する同定基準を超える必要があります。
重要なのは、検索エンジンの結果を盲信してはいけないということです。内容を確認するためにも、タンパク質同定に紐づけられたペプチドのデータを必要に応じてチェックし、ソフトウェアが正しい判断をしたかどうかを確認してください。
最後にMASCOT検索結果の再現性についても言及します。MASCOTの検索結果は常に同じ結果になりますか?という問いについては、「はい、その通りです」とお答えできます。 何がどのように検索されたかを改めて検証したい場合、結果ファイルの中には入力データ、パラメーター、検索時にマッチしたペプチドやタンパク質の情報すべてが含まれています。必要に応じて再検索やパラメーター変更における差異を見るなどといった検証も実行可能です。
Keywords: FDR, interpretation, protein inference, reproducibility, scoring