新しくなった 定量解析レポート[Mascot Distiller 2.8]
Distillerの以前のバージョンにおける定量結果のレポートは、XMLエクスポート(XSLT変換)を使用して処理されていました。XSLTは強力な言語ですが、一般的に使用されていないためレポートをカスタマイズするのが容易ではありませんでした。Mascot Distiller 2.8からレポート機能はスクリプト言語Pythonで記述され、Mascot Parserを使用することができるようになりました。Pythonは一般的に使用されているプログラミング言語であり、Mascot Parserを使用することで、検索結果および定量結果に簡単にアクセスすることができます。
Mascot Distiller 2.8でもこれまでのレポート機能を使い続ける事ができます。Pythonを利用するようになったことでさらに柔軟性が高まり、それに加えて2.8からいくつか追加の出力機能が追加されました。ANOVA、箱ひげ図、クラスタリングのレポート、ボルケーノプロットなどの他、タンパク質・ペプチド定量データをRやPerseusなどで簡単に利用可能なフォーマットでエクスポートする事ができます。
なお現段階(Distiller 2.8.2あるいはそれ以前のバージョン)において、判明したものの未解決な3つのレポートスクリプトのバグがございますのでご注意ください。これらはMascot Distillerのテクニカルサポートページにあるように、ご自身で関連するスクリプトを編集(あるいは弊社にて準備した修正スクリプトとファイルを入れ替え)することで簡単に修正できます。バグは次のリリースでは最初から修正された状態になります。レポート作成
レポート作成はウィザード形式で実行され、作成に必要なパラメーターを指定します。例えば、グラフを以下のいずれかの形式でエクスポートするか選択できます。
- Scalable Vector Graphics(.svg)
- Portable Network Graphics (.png)
- Interactive Javascript
.svg と .png は静止画像ですが、3番目のインタラクティブ Javascriptは plotly という描画ライブラリを用いて作成しており、ズームやツールチップ(カーソルを合わせると補足説明が表示)のような操作が可能です。オプションで、グラフをPlotlyウェブアプリケーションにエクスポートしてさらなる編集や注釈を可能にすることができるなど、論文投稿用の図の作成プロセスを大幅に簡素化することができます。
ANOVA、PCA(主成分分析)、Hierarchical clusteringなどの統計レポートのいくつかは、欠損値がないことが求められますが、欠損値に対して以下のオプションが用意されています。
- 欠損値を含むタンパク質をリストから除去
- 何かしらの固定値に置き換える
- K近傍法を使用した代替値への変換
固定値を入力する、またはK近傍法を使用して値を代入することを選択した場合、欠損値を置換または代入するケースの最大数を設定するように求められます。
レポートでは、コンタミネーションデータベースを指定することもできます。選択したデータベースに含まれるタンパク質は、定量レポートから除外されます。
レポートのカスタマイズ
自身で作成したPythonレポートをDistillerに追加することもできます。レポートは、Mascot Distillerに表示されるウィザードの中身を定義したXMLファイルと、Pythonスクリプトの2つのファイルで構成されています。検索結果や定量結果には、Mascot Parserを使用してアクセスします。Python用のMASCOT Parserは、Distillerで使用されるPythonの組み込みコピーと一緒にインストールされています。レポートを実行できるようにするためには、PythonのソーススクリプトとWizard XMLファイルをC:\ProgramData\Matrix Science\Mascot Distiller\reportsフォルダに置いた後、Mascot Distillerを再起動する必要があります。
既存のカスタムXSLTレポートについては以前のバージョンと同じ使い方、すなわちコマンドラインから、-quantreportスイッチを使用してXSLTテンプレートへのパスを渡して実行する事ができます。コマンドラインでのMascot Distillerの使用方法について、詳しくはMascot Distillerのヘルプに記述がございます。
LFQデータセット PXD026930 を使ったレポートの作成例
Mascot Distiller 2.8に搭載された新しいレポートの一部を紹介するため、PRIDEリポジトリからラベルフリー定量を行ったデータセットを取得して実際にレポート出力を行いました。このデータセットは、酵母(S. cerevisiae)におけるアラニルtRNA合成酵素の役割について調べた論文の関連データです。アミノアシルtRNA合成酵素はヒトの神経疾患と関連する必須酵素です。論文では、酵母において変異がアミノ酸制御経路やヒートショック応答に対して影響を与えることが示唆されています。
実験では、野生型と、アラニルtRNA合成酵素に変異を持つ2つの酵母株(C719AおよびG906D)の3種類を調べています。
培養は30℃で行い、サンプリング後、37℃に上昇させてから2時間経過後再度サンプリングしています。30℃の野生型とG906Dは3回サンプリングしましたが、37℃のC719AとG906Dは2回だけサンプリングし、合計15回の分析を行っています。
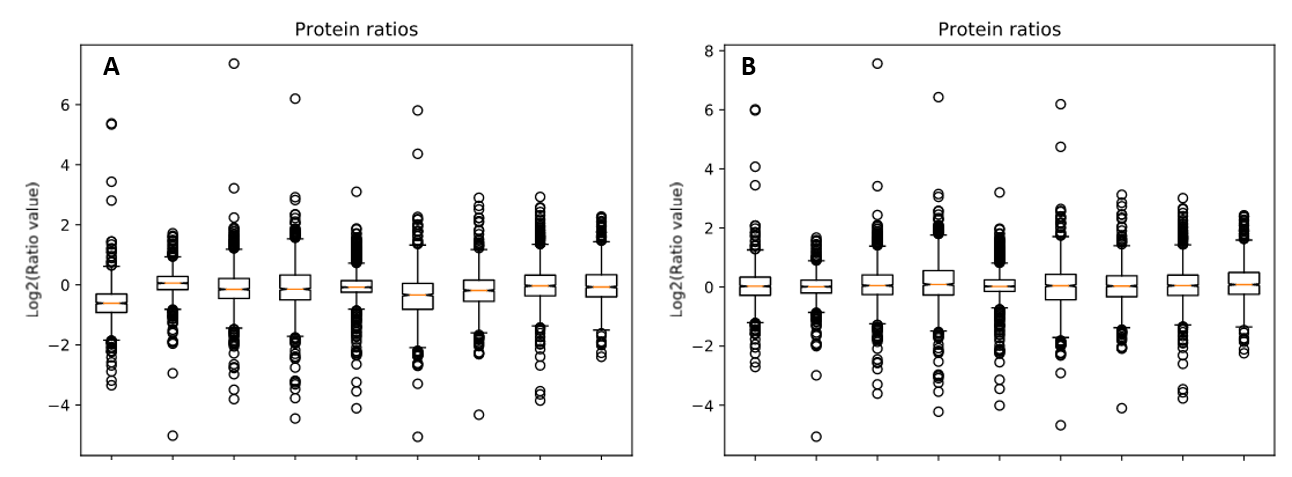
今回このブログ記事の筆者はサイトからrawデータを取得し、Mascot Serrverと Distillerを使って再解析しました。生成されたピークリストをMascot Server 2.8.1で、PRIDEに記載されている検索設定の情報と同じパラメーターで検索し、その後Mascot Distillerで定量計算を行いました。各温度・各変異株について、野生型サンプルに対する各タンパク質の含有比率を求めました。そして解析の最初に、各サンプルにおけるタンパク質比率の中央値を算出しました。各サンプルには同じ量のタンパク質がロードされており、各サンプルのタンパク質比率の中央値は同じ、つまり野生型に対する比率を計算したときに値が1になると考えられるので、比率の中央値を使用してNormalizeする事が可能です。この効果は、「箱ひげ図」レポートを実行することで確認できます。以下の図1は、Normalizationを有効にした場合としなかった場合のタンパク質比のボックスプロットを示しています。
Click to view full size image
図1:タンパク質比の正規化A)を無効、B)を有効にしてボックスプロットしたタンパク質比率の分布を表す箱ひげ図。
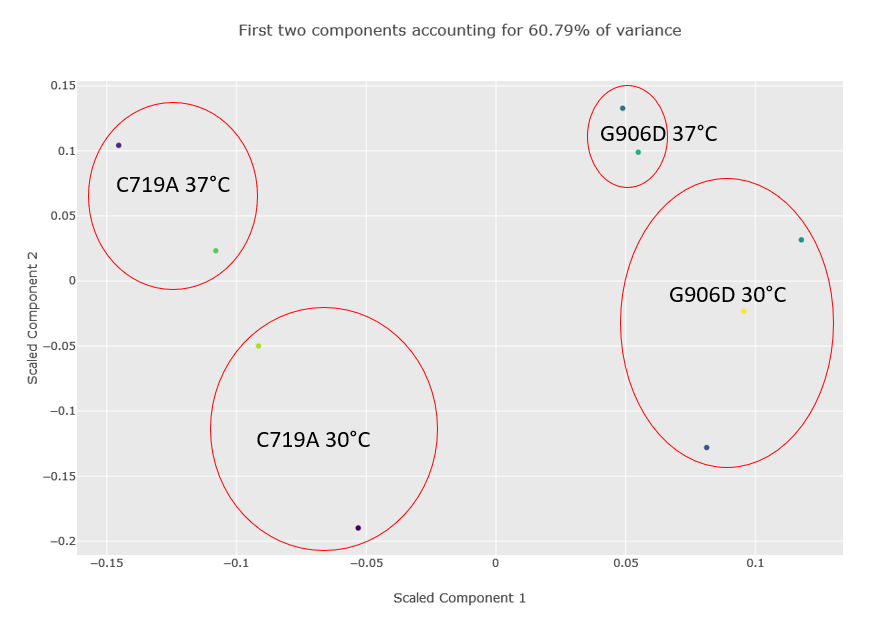
続いて以下の図2は、主成分解析を行った際の第一主成分並びに第二主成分のプロットです。コンタミデータベースのマッチング内容は除外され、レポート実行時に最大2つの欠損値に対して、K近傍法による代入を実施しています。プロットから、第一主成分がC719AサンプルとG906Dサンプルを明確に分離していることがわかります。また第二主成分は、サンプル別に見たときに温度の違いを区別するのに貢献しています。ただし、G906Dの30℃の2サンプルは第二主成分が正の値ですが、他の30℃サンプルはすべて負の値になっています。
Click to view full size image
図2:主成分分析によるプロット。この画像はDistillerが標準出力する内容を編集しており、レポート実行後にグループの分かれ方を示す円(楕円)と、G906Dの外れ値がハイライトされています。
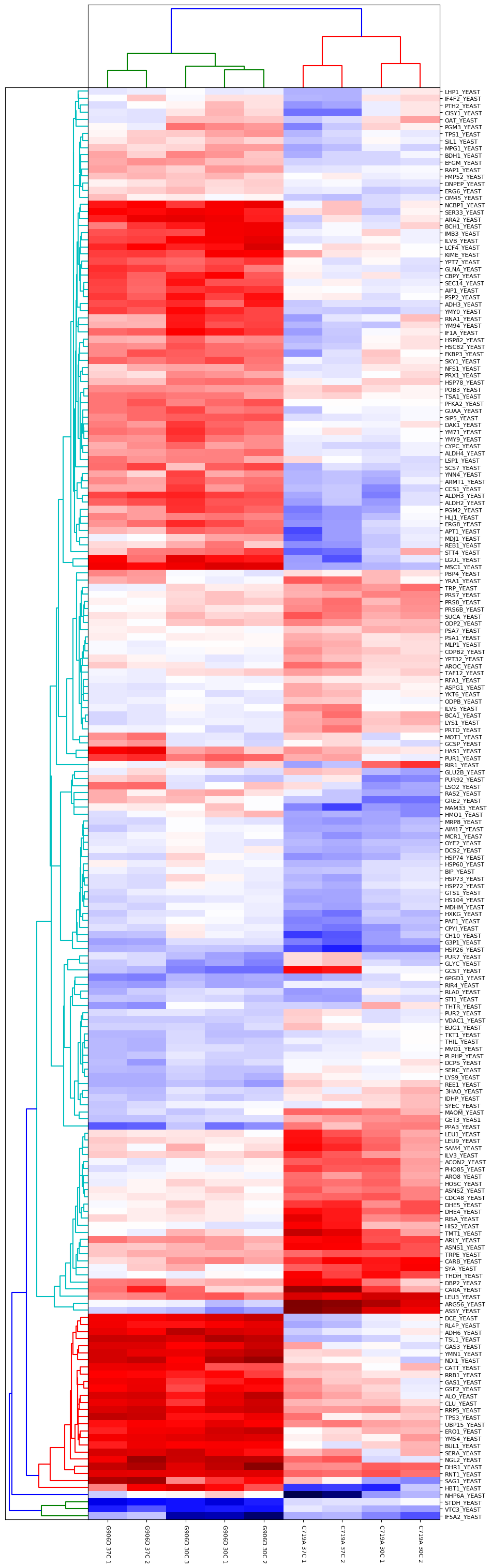
異なる変異株や温度を特徴付けるタンパク質をより広範囲に渡って見るため、階層的クラスタリングレポートを試してみることができます。残念ながら今回のサイズのデータセットでは、野生株と変異株、異なる生育温度の間で本質的に変化しないタンパク質が非常に多く、生成されたデンドログラムやヒートマップで目立った特徴を抽出する事ができませんでした。そこで別のアプローチとしてANOVA検定を実施しました。ANOVAでは異なるサンプルを別々のグループに分けることができます(この場合、2種類の変異株と2種類の生育温度で構成される4つのグループが存在します)。これにより、異なるグループ間およびグループ内で有意に異なるタンパク質を特定することができます。"ANOVA plus clustering" レポートを選択すると、ANOVAの計算結果は、階層的クラスタリング・レポートの実行に使用されます。このレポートから生成されたデンドログラムとヒートマップが下記の図3です。
Click to view full size image
図3:” ANOVA plus clustering report ”の出力。4つのグループ(G906D 30℃, G906D 37℃, C719A 30℃, C719A 37℃)を定義し、有意水準5%としました。算出されたp値はBenjamini-Hochberg法を用いて多重検定用に補正済。欠損値はK-近傍法を用いて代入(上限2つまで)。
図3からわかるように、2つの変異体はいくつかのタンパク質グループの挙動にはっきりとした違いがあります。例えば、アルコール脱水素酵素3型など様々な代謝経路に関与する酵素について、野生型と比較して30℃と37℃の両方でG906Dでは発現量が大きく上昇するのに対し、C719Aでは逆に大きく低下するといった違いが見られます。
同じ変異型において、異なる生育温度間の差異も存在します。例えば、LHP1_YEAST、IF4F2_YEAST、PTH2_YEAST、CISY1_YEASTといったタンパク質については、30℃と37℃で培養したC719Aで明確な違いがあります。
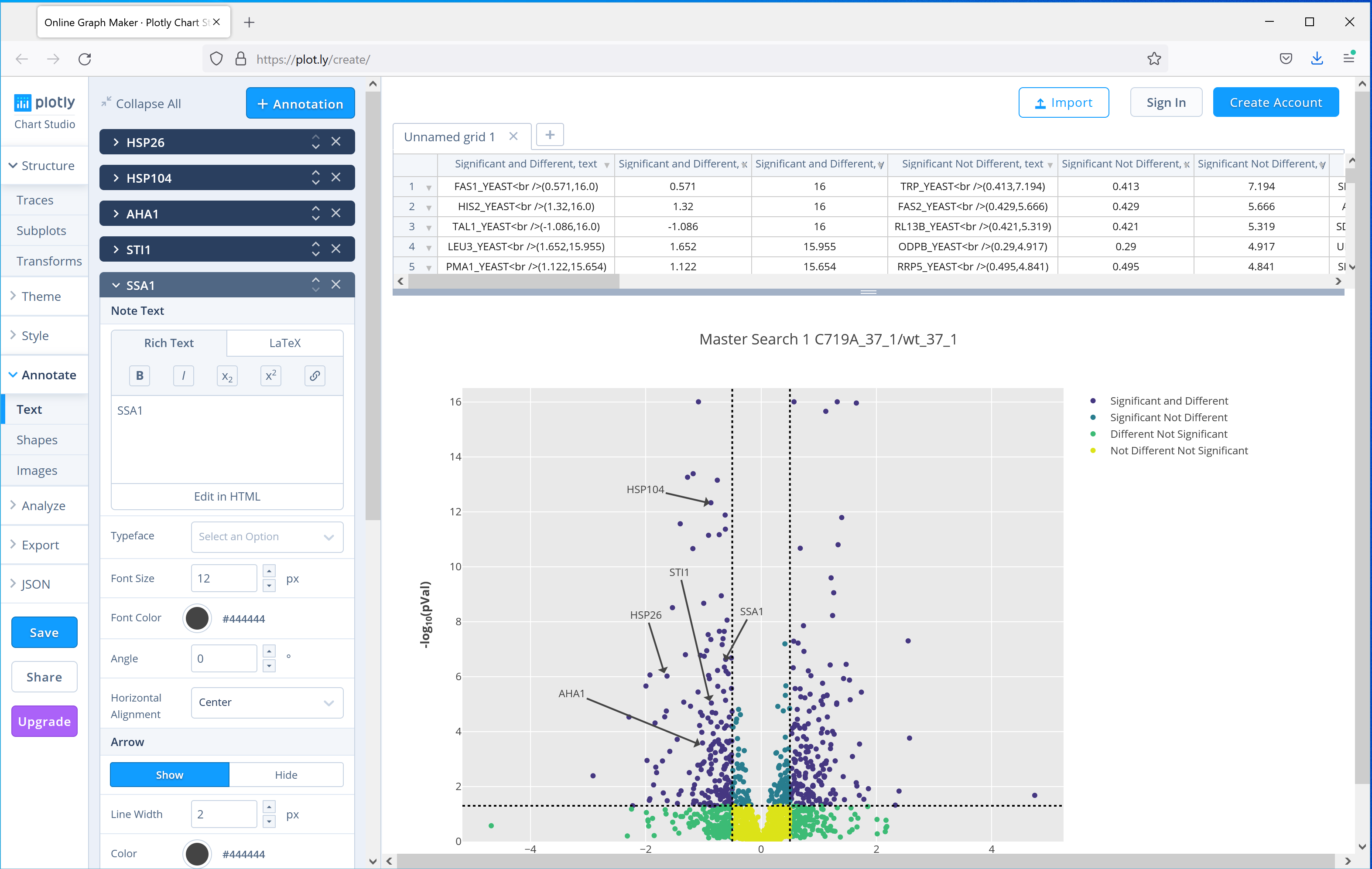
Distiller 2.8では、ボルケーノプロットの表示も可能であり、発現量が有意なレベルで増加または減少したタンパク質がわかりやすく表示されます。論文では、37℃で培養したC719A変異体の結果を野生型と比較したボルケーノプロットを用いて、変異体で発現量が低下している同じパスウェイの5つのタンパク質に注目しています。図4はボルケーノプロットです。プロットの各データポイントにタンパク質のアクセッションのラベルを追加し、さらにサードパーティのplot.lyウェブアプリケーションでプロットを開いてグラフに注釈を付けるオプションを選択しています。このように、これらのツールを使うと論文などで使用可能な図を簡単に作ることができます。
Click to view full size image
図4:37℃で培養した野生型とC719A変異体のタンパク質発現量を比較したVolcano plotレポート。図はplot.lyウェブアプリケーションにアップロードされ、さらなるアノテーションを行っています。論文で取り上げられた5つのタンパク質についてハイライトしています。
Mascot Distiller 2.8に含まれているPythonレポートは、定量データに対する一般的な分析を幅広く提供し、簡単に論文発表用の図の作成を行う事ができます。すでにMascot Distillerのライセンスをお持ちの方は、2.8へのアップデートは無料です。Mascot Distillerのライセンスをお持ちでない方は、30日間の試用が可能です。詳しくはMascot Distillerのダウンロードページをご覧ください。
Keywords: Mascot Distiller, Python, quantitation, reporting, XSLT