Mascot Distillerによるインタクトなクロスリンクスペクトルデータのピーク抽出
インタクトなクロスリンクデータセットのピーク抽出を行う際、いくつかの点で注意が必要です。例えばペプチドの架橋結合によりプリカーサーの質量が大きくなり、より多価になる傾向がある事、MS/MSスペクトルがアルファおよびベータペプチドからのフラグメントを含むキメラスペクトルとなっていて非常に複雑である事、などが挙げられます。 今回このブログ記事では、ピーク抽出に関するパラメータの選択と効果を確認するため、過去のブログ記事「クロスリンクペプチドの検索結果検証」でも使用した公開データのrawファイルを使って色々と試しています。
フラグメントイオンの電荷状態
Mascot ServerではMS/MSマッチングにおいて、1+と2+のフラグメントイオンしか検索しません。クロスリンクペプチドのデータセットのように多価のプレカーサーがある場合、生成されるフラグメントイオンの多くも同様に高い電荷状態になり、3価以上のピークはそのままではマッチングできません。最悪、正解ではない別の結果が正解として提示される可能性もあります。 このようなケースでは、フラグメントイオンのピークを1価相当の値に変換(MASCOT DistillerではDechargeと記載)するか、Mascot 2.7以降をご利用であればmgfファイルの各ピーク行に電荷情報を付け加えて対処をする事をお勧めしています。それらの対応はMascot Distillerでも可能ですが、MS/MSスキャンがプロファイルではなくセントロイドとして保存されている時はデータをアンセントロイド化を使用する必要があるのでご注意ください。
Mascot Distiller 2.8.3にて、以下の処理設定でピークピッキングを実施しました。
- default.ThermoXcalibur.opt設定ファイルを使用。価数をDistiller上で決定せず、rawファイルから取得したセントロイドピーク値をそのまま出力。
- 「クロスリンクペプチドの検索結果検証」で使用されている設定ファイルを使用。すなわち1ダルトンにつき100ポイント (ppDa) の分解能で MS/MS スキャンをアンセントロイド化。
- prof_prof.ThermoXcalbur.opt設定ファイルを使用。600ppDaの分解能でMS/MSスキャンをアンセントロイド化。
ピークリスト作成後、Mascot Server 2.8.2を用いて検索しました。 defaultファイル以外の解析については、MS/MSスキャンデータではアンセントロイド化され、フラグメントイオンの価数をDistiller上で特定し、フラグメントイオンのピークをm/zではなく1価相当に換算した値に変換して出力するオプションを選択しました(Mascot Distillerの環境設定ダイアログの「Peak list format」オプション)。 結果は以下の表1に示すとおりです。
| Processing options | Uncentroiding points per Da | Decharged fragment ions? | Time taken (mm:ss) | Significant CSMs (p<0.05) |
|---|---|---|---|---|
| default.ThermoXcalibur.opt | N/A | No | 00:14 | 655 |
| Previous blog settings | 100 | Yes | 02:34 | 1087 |
| prof_prof.ThermoXcalibur.opt | 600 | Yes | 12:08 | 1143 |
ひとつ注目すべき点があります。前回のブログ記事の処理オプションを使って結果を比較した場合、クロスリンクペプチドマッチ数(CSMs)が増えています – 前回1008でしたが今回は1087です。 前回はMascot 2.7を使用してデータを検索していましたが、今回利用したMascot 2.8ではクロスリンクペプチドの検索にいくつかの改良が加えら、さらに79件のマッチングが得られました。
default.ThermoXcalibur.optの結果はアンセントロイド化した場合よりも大幅に悪くなります。 これは2価を超えるフラグメントイオンを含むピークリストが多く、それらのピークはピークリスト中のフラグメントイオンをdechargeしない限りMascotでマッチングされないからです。 例えば下の図1は、default.ThermoXcalibur.optとprof_prof.ThermoXcalibur.optのピーク処理設定を使用して4+の同じクロスリンクペプチドに対するマッチング結果を比較したものです。
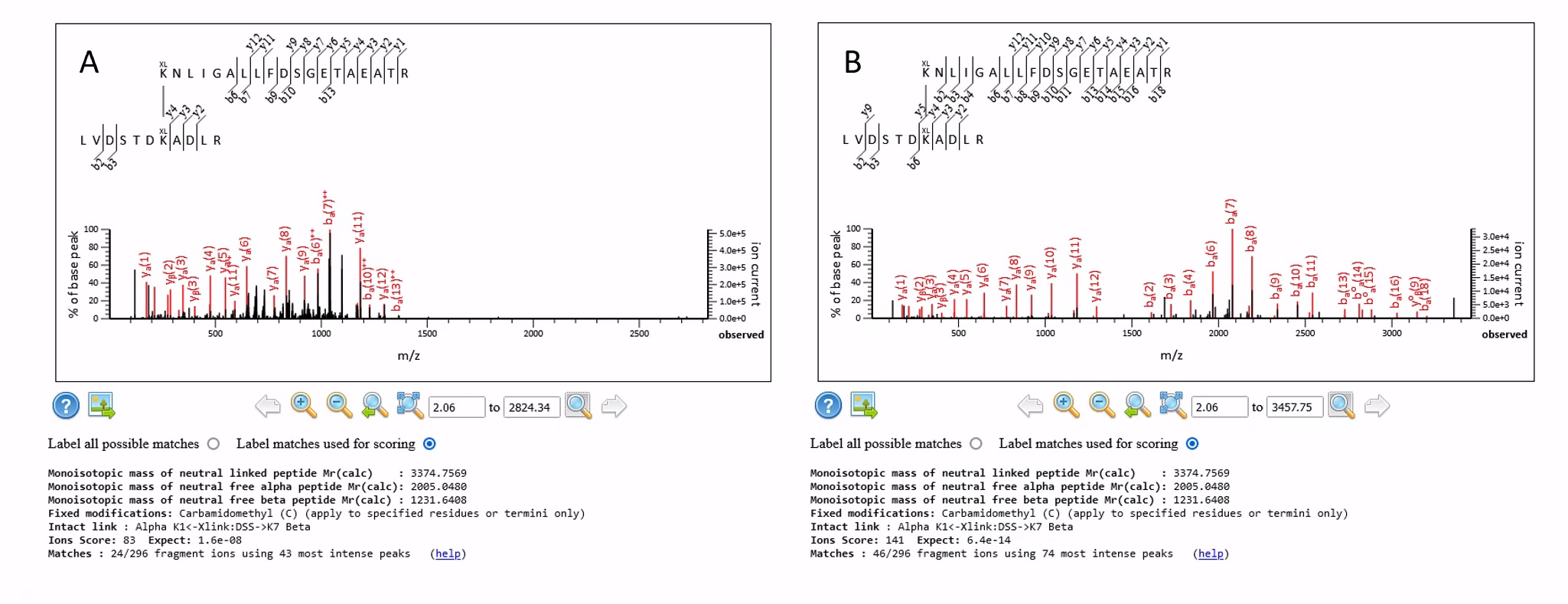 図1:A) default.ThermoXcalibur.opt と B) prof_prof.ThermoXcalibur.opt を用いてデータを処理した後、decharge(1価換算)させた、同じクロスリンクペプチドに対するマッチングの比較
図1:A) default.ThermoXcalibur.opt と B) prof_prof.ThermoXcalibur.opt を用いてデータを処理した後、decharge(1価換算)させた、同じクロスリンクペプチドに対するマッチングの比較
どちらも良いマッチングですが、3+フラグメントイオンがそれなりの数(と4+が少し)あることがわかります。
prof_prof.ThermoXcalibur.opt設定によりdechargeを行ったピークリストは3価以上のフラグメントもマッチングされ、αとβペプチドの両方のカバー率が上がります。フラグメントイオンのカバー率は24から46となり、スコアも83から141に向上しています。
アンセントロイド化の解像度(ppDa)
Prof_prof.ThermoXcalibur.opt の設定の適用は前回のブログ記事で使用した設定よりも優れており、56個多く有意なCSMsを検出しましたが、処理時間という大きな代償を払うことになりました(約5倍の時間がかかりました)。以前のブログ記事「Defaultとprof_prof どちらが良いか : Mascot Distillerを用いたThermo .RAWデータのピークピッキング処理オプション(英語版、日本語版)」において、prof_profオプションのuncentroiding設定を200または400 ppDaに下げた場合、処理時間を大幅に改善しながらも良い結果が得られることを実証しましたので、今回のクロスリンクデータでも同じことを試みてみました。 結果は以下の表2にまとめられています。
| Uncentroiding ppDa | Time take (mm:ss) | Significant CSMs (p<0.05) |
|---|---|---|
| 200 | 02:33 | 1139 |
| 400 | 06:25 | 1141 |
| 600 | 12:08 | 1143 |
ご覧のように、200ppDaあるいは400ppDaのどちらを使ってもデフォルトの600ppDaに近い結果が得られた一方、処理時間は約1/5あるいは1/2になりました。 高い解像度でアンセントロイド化するとピークがマージされるリスクが減りピーク抽出改善されます。そのため600 ppDaでアンセントロイド化した場合、200または400 ppDaで行った場合と比較して少しだけ多いCSMが見つかりますが、そのわずかなメリットのために非常に長い処理時間がかかっている事になります。この結果から、処理速度と結果の質のトレードオフとして合理的な基準は400ppDaであると思われます。なおデータがセントロイドではなくプロファイルとしてデータを保存している場合、このトレードオフの話は関係ありません。
S/N
一般的にピークリストのS/Nが良好であれば、データベース検索でより良い結果を生み出します。 インタクトなクロスリンクペプチドからのMS/MSスペクトルは複雑なキメラスペクトルであるため、ピークリストのS/Nがより重要になります。 図1より、Mascot Distillerでピークピッキングを行うことで、単純にセントロイドデータを使いうよりもS/Nの良いピークリストが得られることが分かります。そこで、Mascot DistillerのMS/MS Peak Pickingオプションタブで "Minimum signal to noise (S/N)" の値を2に上げ、400 ppDaでアンセントロイド化し、ピークリストの再処理を行いました。 結果は表3にまとめられています。
| Uncentroiding ppDa | Minimum signal to noise | Time taken (mm:ss) | Significant CSMs (p<0.05) |
|---|---|---|---|
| 400 | 1 | 06:25 | 1141 |
| 400 | 2 | 05:24 | 1152 |
これらのデータでは、S/N比を上げることで11個のCSMsが追加され、処理時間が1/6に短縮されました。こちらはパラメータの変更が検索速度と結果の両方に良い結果をもたらしたことになります。ただし高すぎるしきい値を設定してしまうと、正しいフラグメントイオンがピークリストから除外されデータベースの検索結果が悪くなりますので注意が必要です。
実際の解析の前に標準的なサンプルを使ったトライアルを行い、今回ご提案したようなパラメータについて変更をしながら同定数と処理時間の最適な組み合わせを探し出した上で、それをご利用のデフォルト設定として標準データに適用して頂く事をお勧めします。
なおこのブログでは、MS/MSスキャンがセントロイドとして保存されるThermo社製の装置からのデータを使用しています。 Thermo社以外の多くの装置では、プロファイルモードでデータを収集するように設定されているので、セントロイドを解除する必要はありませんが、フラグメントイオンの1価換算とS/N比の調整については、今回の記事の知見が役立ちます。
Mascot Distillerをお持ちでない方でご自身のデータで評価したい場合、Mascot Distillerの30日間トライアルを提供しています。詳しくは弊社までお尋ねください。
Keywords: crosslink, crosslinking, Mascot Distiller, peak picking, Thermo