DIA-MSでの使用を目的とした高品質なスペクトルライブラリーの作成
MASCOT Server,Daemon,Distillerを駆使してスペクトルライブラリーを作成する
Mandaらによる最近の論文[1]では、Data-Independent Acquisition(DIA)データの検索に使用する事を目的として、DDAデータから高品質のスペクトルライブラリーを生成するためのパイプラインについて説明しています。このパイプラインはDIAデータ解析に適したライブラリーを生成するために様々なツールの集合体から構成されており、rawデータから目的のライブラリーを作成する事ができます。
Mascot Server 2.6では、NIST MSPepSearchを使用したスペクトルライブラリーの検索機能を導入しました。 また関連する機能として、Mascotの検索結果からスペクトルライブラリーを生成する機能も搭載されました。生成されるライブラリーのフォーマットはNIST MSPフォーマットで、DIA-NNやSkylineなどのDIAの検索ツールで利用可能です(訳者注:Scaffold DIAにはMSPからDLIBフォーマットに変換するツールが搭載されています)。
本ブログでは、Mandaらが使用したデータセットを用いて、Mascot DistillerとMascot Daemonでrawデータファイルを処理し、Mascot Serverのスペクトルライブラリー生成機能でMSP形式のファイルを作成する方法をご紹介します。該当データであるHEK293細胞株のデータセットはこちらからダウンロードできます(9.3GB)。
DIA検索においてin silico予測ツールのモデルに含まれない様々なvariable modificationを考慮した検索を行う場合、現状ではDDA検索結果から作成したスペクトルライブラリーを使用する方法が主流となっています。またMascotを使用するメリットとして挙げられるのが、ETDフラグメンテーションを使用しているケースです。 MSPフォーマットはa-、b-、y-イオンしかサポートしていませんが、Mascotはc-、x-、z-イオンを適切なニュートラルロスを持つb-、y-イオンとしてエクスポートします。
ステップ1:データセットに適した検索パラメーターを決定
Mandaらの論文でも使用した検索エンジンの1つにMASCOTも含まれていました。そのため検索パラメーターの設定は論文のsupplementary dataで確認する事もできます。しかしよりよい結果を得るため、スペクトルライブラリー作成に適切なパラメーターの決定方法はこのブログ記事の手順を適用しました。すなわちMascotのError Tolerant Searchを使用して数多く検出される修飾を見つけ、これを改めて指定して検索を行い、スペクトルライブラリーを得ます。まずデータセットからraw ファイル1つを選択し、Mascot Distillerを使用してデータを処理し、Mascot Server 2.8に対して以下の検索設定でError Tolerant Searchを実施しました。(データベース:UniProt Human proteome、Decoy検索も実施)
| Target FDR | 1% |
| Enzyme | Trypsin |
| Fixed modifications | Carbamidomethyl (C) |
| Peptide mass tolerance | 25 ppm |
| Fragment mass tolerance | 0.05 Da |
システインのカルバミドメチル化を固定の修飾として設定していますが、可変の修飾はなにも指定していません。検索後、結果画面のヘッダー領域に表示される同定された修飾の一覧リストを見て、検索で指定するべき修飾を決定しました。選択された内容は以下の通りです。
| Modification | Delta | Type | Site | Total matches |
|---|---|---|---|---|
| Carbamidomethyl | 57.021464 | fixed | C | 2419 |
| Non-specific cleavage | ET | - | 351 | |
| Guanidinyl | 42.021792 | ET | N-term | 139 |
| Carbamidomethyl | 57.021464 | ET | M | 118 |
| Deamidated | 0.984016 | ET | N | 110 |
| Carbamidomethyl | 57.021464 | ET | N-term | 74 |
| Oxidation | 15.994915 | ET | M | 55 |
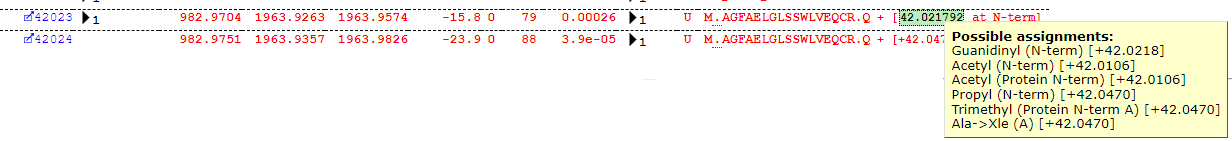
この結果に対しては少し補足説明が必要です。 ほとんどの修飾は問題ありませんが、Guanidinylは+42Daの候補の中で最適とは言えないかもしれません。Error Tolerant Searchの結果画面では、質量差と対象のアミノ酸残基に対して修飾の可能性が複数あった場合そのすべてが表示されますが、この表にまとめられる際は見やすさを重視するため質量差がほぼ同じ修飾候補リストの中で最初にリストアップされたものしか報告しません。プラス1Daのジレンマと同様、プラス42Daも値が非常に近い質量の範囲にいくつもの修飾候補があります。例えばタンパク質のN末端のアセチル化などが挙げられます。 実際にいくつかのマッチを詳しく見てみると、以下図のようにタンパク質のN-末端にマッチするものが多いため、おそらく最も多い修飾はタンパク質N末端のアセチル化であることがわかります。
修飾の可能性に加え、Error Tolerant Searchによって特定された非特異的な切断マッチが大量にあり、これは元の論文では扱われていませんでした。この結果を踏まえ今回は非特異的な切断についても検索時に考慮する事にしました。
ステップ2:多くのVariable modification指定と非特異的な切断パターンを考慮して検索を実施
Step1の検証を元に2種類の検索を実施しました。1つは切断パターンが通常のトリプシン設定、もう1つは最初の検索でヒットしなかったデータに対して、切断パターンをセミトリプシンにした検索を行っています。どちらの場合もVariable modificationとして、Deaminated (NQ), Acetyl (Protein N-term), Carbamidomethyl (M), Carbamidomethyl (N-term), Oxidation (M) を指定し、FDR 1%、コンタミデータベースとUniprot human proteome データベースに対して検索を行いました。なおここでご紹介した2段階の検索は、Mascot Daemonを使って実現可能です。まず先に2回目の検索の方、セミトリプシン設定の検索を「フォローアップ検索」として作成します。その後、1回目に行うトリプシン設定の検索を作成します。 この時、期待値が0.01未満のペプチドを、策に設定したフォローアップ検索のタスクに渡すよう指定します。どちらの検索もMascot Distillerを使用してrawデータのピーク検出処理を行っています。検索にDaemonを使用することで、2つの検索の検索タイトルに共通部分や識別するための別の字文字列をつける事で、後のプロセスでスペクトルライブラリーを生成する際データ処理がよりスムーズになります。
検索を実行する際、Mascot ServerはMS/MSの理論フラグメントとして1+と2+のイオンシリーズのみ準備をするという点にご注意ください。3価以上のフラグメントイオンが存在する場合、それらはそのままではマッチングに利用されません。 それらのピークもマッチングに利用するためにはdecharge(すべてのピークのm/zを1価相当の値に換算して出力)する必要がありますが、そうすると生成されたライブラリーもすべて1価換算されたm/z出力になってしまうため、MascotやSkyline、DIA-NNなどの外部ツールでライブラリーを使用する際にその点について気を付けてください。
ステップ3:1%のペプチドFDRとなるような期待値を計算
スペクトルライブラリーには信頼できる結果のみを含むのが好ましいです。そこでライブラリーを作成する際の基準としては1%のFDRを満たす期待値を閾値として指定します。 Mascot Daemonでは複数検索内容を結合した結果画面を開く事ができます。そしてさらに結合後の結果画面内の設定を使用して、1%のPSM FDRを満たすように期待値の閾値を調整する事ができます。
(公開された論文とは異なり、より新しいバージョンの)Mascot 2.8では検索の最初の段階でFDR 1%を閾値とするように設定できます。上記の操作により、閾値となる期待値は以下のようになりました:
- トリプシン検索の閾値0.00679
- セミトリプシンの検索閾値:0.001721
ステップ4:ライブラリーの作成
2つの検索において閾値が大きく異なったため、トリプシン検索結果とセミトリプシン検索結果についてはそれぞれライブラリーを分けて作成することにしました。
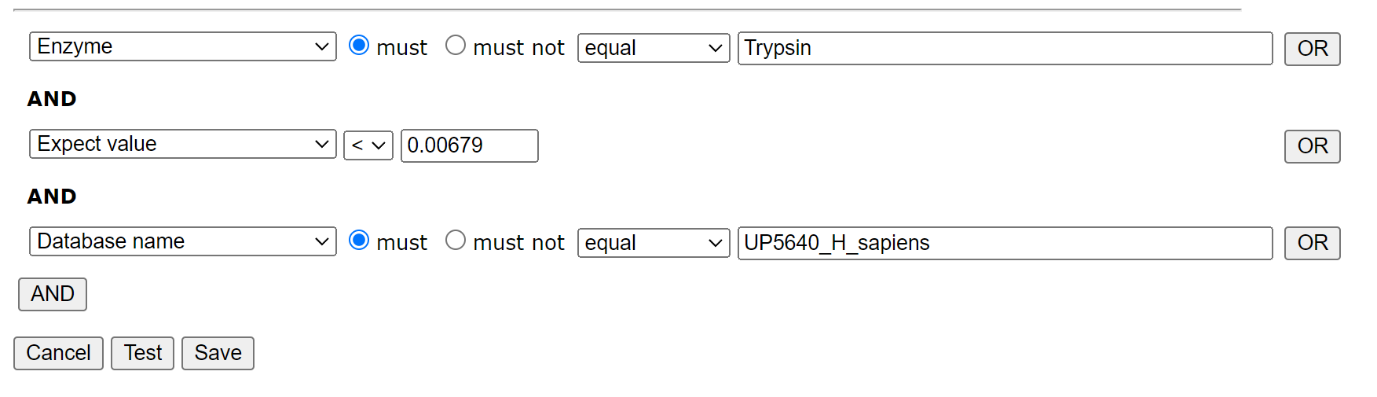
MASCOTのDatabase Manager 機能を経て新しいライブラリーを作成するには、左フレームの「Library」→「Create new」を選択します。ほとんどの場合、関連する設定値はデフォルトの値で問題ありませんが、適切な参照データベース(訳者注:Accession情報をもとにタンパク質の配列を探すためのデータベース)を選択する必要があります。それらの設定が終わったら、Edit filtersを選択してください。 繰り返しになりますが、ライブラリーはトリプシン検索の結果とセミトリプシン検索の内容を分けて作成します。 切断パターン(酵素設定)、データベース、期待値でフィルターをかけ、さらにコンタミデータベースにマッチした内容を除外したライブラリーを作成します。
HEK_semiTrypsin ライブラリーのためのフィルター: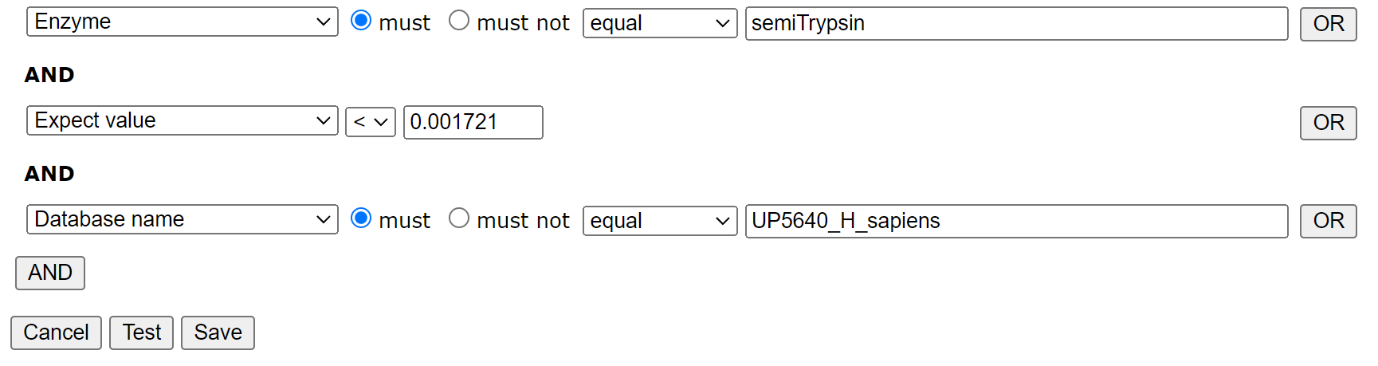
フィルター作成後元の画面に戻り、Import search resultボタンを選択してライブラリー作成を行います。今回ご紹介した例と異なり結果の選択についてのフィルターリング条件が難しい場合、まとめたい結果ファイル(.datファイル)をすべて適当に作成したフォルダーにコピーし、対象選択を単純なワイルドカードパスで一度にすべて選択するのが最もシンプルな操作となります。今回の場合、ステップ2でトリプシン検索とセミトリプシン検索を区別する目的で、共通項目あるいは逆に両者を区別する情報である検索タイトルを定義しているため、検索日付とタイトル情報を使ってフィルターをかけるのが比較的容易な状況でした。
ステップ5:ライブラリーを利用する
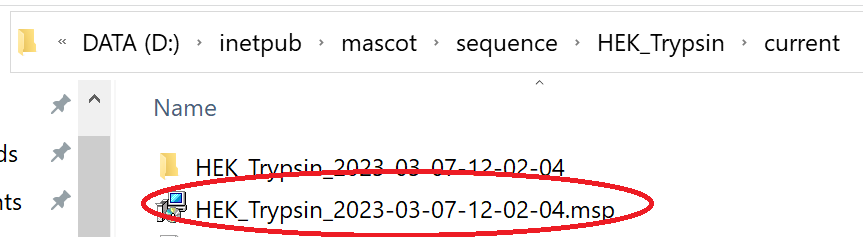
この操作を経て、Mascot Serverでライブラリーを使用できるようになりました。また下図のようにMascot Serverのsequenceディレクトリの下にはMSPファイルが作成されました。
ライブラリーのcurrentディレクトリからMSPファイルをコピーし、SkylineやDIA-NNなどのDIA解析プログラムでご利用ください。 さらにDatabase Managerは、ライブラリー作成の際にインポートされたもの、されなかったものを正確に記録した完全なログが出力します。 これらのログファイルはライブラリーの「incoming」ディレクトリに保存されます。
参考文献
1 Srikanth S. Manda, Zainab Noor, Peter G. Hains, and Qing Zhong, PIONEER: Pipeline for Generating High-Quality Spectral Libraries for DIA-MS Data. Current Protocols, 1, e69. doi: 10.1002/cpz1.69
Keywords: DIA, Mascot Daemon, Mascot Distiller, Mascot Server, spectral library