キメラスペクトルのペプチド同定(DDA、DIA)
プロテオミクスの分野で昔から利用されている多くの検索エンジンでは、ペプチドのMS/MSスペクトルが単一のプリカーサーペプチド由来で生成されることを前提としています。これは狭い分離ウィンドウ、Data Dependent Acquisition(DDA)モードで1つのトリプシン消化ペプチドをとりだしフラグメンテーションさせてデータ解析する場合に当てはまっていた方法です。これに対してMascot Serverではversion 2.5より、複数のプリカーサーペプチドが混合したMS/MSスペクトル、キメラスペクトル(Chimeric Spectra)のペプチド同定をサポートしています。しかし残念ながらこの機能は皆様にあまり知られていないようです。今回はキメラスペクトル解析に関する小技をご紹介します。
キメラ・スペクトルデータの検索
ピークリストをMascot Serverに検索させる際、多くのケースでMascot Generic Format(MGF)フォーマットが利用されています。このフォーマットの基本的な形式は以下の通りです:
BEGIN IONS TITLE=Spectrum 2 PEPMASS=896.05 25674.3 3+ SCANS=123 345.10 237 ... END IONS
このうちPEPMASS行で指定している情報が意味するのは、プリカーサーのm/z (896.05)、強度(25674.3)、電荷(3+)です。複数のプリカーサーが開裂しフラグメントピークが1つのスペクトル内に混在している事が明らかなケースのデータに対してもMGFフォーマットは対応しています。1つだけでなく、複数のPEPMASS行を追加するだけです:
BEGIN IONS TITLE=Spectrum 2 PEPMASS=896.05 25674.3 3+ PEPMASS=896.43 1084.9 3+ SCANS=123 345.10 237 ... END IONS
ただしPEPMASS行を追加させたデータを実際に作成可能かどうかはピークリスト作成ソフトウェア次第です。もし現在ご利用のピーク抽出ソフトウェアではピークリストに複数のPEPMASSラインを含むMGFファイルを生成できない場合、Mascot Distillerであれば可能ですので是非お試しください。
Mascotがキメラスペクトルのクエリーを読み込む際、後述するようなキメラスペクトルデータ特有の特殊な処理を行うため以下の2つの処理を行います。それは、1)それぞれのPEPMASS行に対して同じピークリストの情報を持たせた2つの補助クエリーを作成し、2)Mascotはソースインデックスと呼ばれる実行中のインデックスを使用して補助クエリーどうしをリンク、という処理です。この準備を完了後にデータベース検索が実行されます。
データベース検索中、それぞれの補助クエリーに対してマッチングとスコアリングが行われます。この時Mascotは、一方の補助クエリーでマッチしたとみなされたフラグメントピークについて、もう一方の補助クエリーでは説明できないノイズとして扱います。Mascotがこれらの自分ではないフラグメントピークについて、単純に除去やピーク減衰をしていないのがポイントです。もしそうしてしまうとフラグメントピークがシェアされているケースにおいてはかえって同定しづらくなってしまい、正しい結果が導き出せない可能性があるためです。
検索が終了すると、Mascotはキメラスペクトルのデータのソースインデックスを結果ファイルのクエリー情報がまとめられたセクションに記録します。ソースインデックスの内容はデータ処理下流のツールにおいてキメラスペクトルを参照する際に使用されます。上記で紹介した内容以外にキメラスペクトルに特化した他の処理は現在のところ行っていません。ピーク抽出ソフトウェアがMGFファイルのSCANS行にスキャン番号を書き込んでいる場合(Mascot Distillerでは行われています)、キメラマッチのグループ化にそのスキャン番号を利用します。書き込まれていない場合はソースインデックスの情報に遡って利用する事でキメラマッチのグループ化を行います。
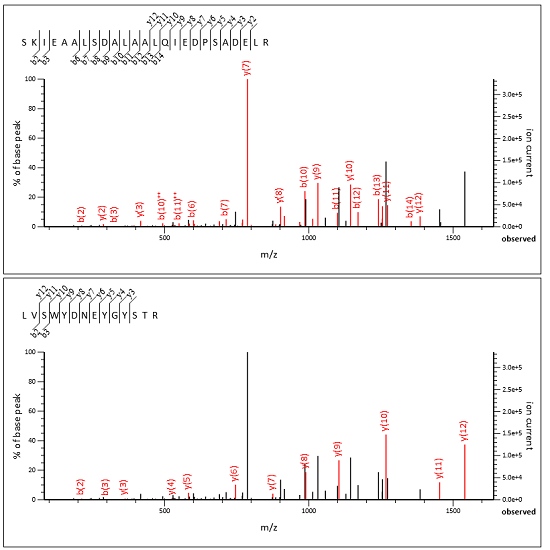
私たちが新しく追加したヘルプページ「Chimeric Spectra」には、キメラスペクトル解析の背景とその詳細が記載されています。下図はそのヘルプページ内で例として使用した、酵母DDAデータセットから取り出したキメラスペクトルのマッチング図です。2つのペプチド由来のフラグメントによって主なピークがアサインされている事が確認できます。
キメラスペクトルに対するMascot 検索:利点と限界
キメラスペクトルに対するMascotアプローチには、そのシンプルさにもかかわらず(あるいはシンプルだからこそ!)利点があると私たちは考えています。その利点とは、良いスコアを得るために最も重要な事が「良いマッチング」であり、同定のために明確なMS/MSマッチングの根拠が必要である、ということです。保持時間やイオンモビリティーのようなスペクトル以外の要因に関係なく、スペクトルを確認しそのペプチドが本当にそこにあると確信できなければならないと考えています。実測と理論のスペクトルマッチングが良くなければ、ソフトウェアが正しく動作しているかどうか、よくわかりません。そもそもこの手法ではなぜMS/MSスペクトルを取得するのか?というところに行きつきます。
しかし裏を返せばこの手法は複雑なデータには弱いと言えます。Mascotのスコアリングはノイズに対して寛容であるものの、プリカーサーの数が増え複雑性が増すにつれて良いマッチングを得ることが難しくなってしまいます。Mascotのキメラスペクトル解析は通常、一次(最も強いピークを生み出す、の意味)と二次(一次の次に強い)のペプチドまでは同定できます。時には3番目や4番目のペプチドを同定できることもあるかもしれませんが、多くのプリカーサーのフラグメントを追加されるにつれ、強度の弱いペプチドのスコアはゼロに近づき、ほとんど同定できなくなります。
TIPs:キメラスペクトル検索結果のレポートスクリプト
Mascot Server の現バージョンのレポートでは、キメラスペクトルデータに対するマッチへの表示が最適化しているとは言えません。私たちはその点に対する応急処置として、キメラスペクトルデータ解析用の補助レポートスクリプトを作成しました。どのバージョンの Mascot でもダウンロードして使用できます。このスクリプトはピークリストに含まれるスペクトルごとのプリカーサーの数をまとめ、さらにその中で同定基準を超えたデータ数も表示する事ができます。
TIPs:Decoyデータベースは逆向きでなくランダムを使う
私たちは、FDR(False Discovery Rate)を推定するため、さらにはPercolatorの再スコアリングを可能にするために、Target-Decoy検索を常に実行するよう推奨しています。しかし、Percolatorが正しく機能するためには、Decoyデータベースは何でもいいというわけにはいきません。Decoyデータベースのスコア分布が、Targetのfalse(またはnull)マッチのスコア分布として正しく代替できているかを考える必要があります。キメラスペクトルデータに対するDecoyデータベースの適用が不適切で、Decoyデータベースの検索結果のスコア分布がTargetのfalseマッチのスコア分布よりも低い方向へ推移してしまうと、FDRは過小評価となってしまい、結果的に不当に同定基準を超えてしまうペプチドが増えます。質の悪いデコイデータベースを使った検索の結果をPercolatorに与えてしまうのは大きな問題なのです。
MascotのDecoy検索機能は、デフォルトで逆向き配列のタンパク質配列を作成するよう設定されています(訳者注:バージョンによります。気になる方は結果画面のFDR表示個所をクリックし、表示される内容をご確認して下さい)。筆者が多くのキメラスペクトルのデータセットについて調査した結果、キメラスペクトルのようにスペクトルの複雑さが増すケースでは逆配列のDecoyデータベースの場合マッチングに苦労することがわかりました。 従って、キメラスペクトルの解析を行うときには、ランダム配列によりDecoyデータベースを作成する事をお勧めします。Mascot Serverのオプション設定で、DecoyTypeSpecificを1(逆配列)から3(ランダム)に変更する事で可能です。
簡単ではありますが、両者の結果の比較をしました。酵母のDDAのデータセットについて、逆向き配列とランダム配列のDecoyデータベースを作成した場合の結果を比較したのが以下の表です。
| Decoy type | Sig. threshold | Target PSMs | Decoy PSMs | FDR |
|---|---|---|---|---|
| reverse | 0.05 | 45506 | 179 | 0.39% |
| random | 0.05 | 45506 | 341 | 0.75% |
reverseの方が良い結果に、即ちFDRの値が低く見えますよね。FDRが低くなるのは、このデータセットに多く含まれるキメラスペクトルに対して、Decoyデータベースとのマッチは軒並みスコアが低いためです(訳者注:Decoyのスコアが不当に低すぎて、結果的にFDRの値が良くなっている、という意味です)。Decoyデータベースの配列をランダムのパターンに変えて検索を行った場合、ターゲット側の同定データ数は変わりませんが、FDRの推定値はより正しい値に近づきます。ただしこのランダムなDecoyデータベースの結果も完全には満足できるものとは言えず、将来のリリースでは改善したいと考えています。
TIPs: DIAデータ
DIA (Data Independent Acquisition)では、分離ウィンドウがDDAより広く、強度ベースのプリカーサー選択はありません。質量範囲内のすべてのプリカーサーが同時にフラグメンテーションされ、キメラ性の高いMS/MSスペクトルが得られます。
DIAの測定アプローチは、選択された分離ウィンドウの幅に基づき、ナローウィンドウ(8 m/zまで)とワイドウィンドウ(> 8 m/z)に大別することができます。 Mascot Serverは現在、スペクトルあたり数十の前駆体を持つことができるようなワイドウィンドウのDIAデータを処理する有効な方法はできません。
しかし、ナローウィンドウのDIAスペクトルであれば、スペクトルあたり数個の前駆体しか持たない傾向があり、複雑さはこれまで説明をしてきたキメラDDAスペクトルと似ているため、Mascotを適用する事ができます。 次回のブログ記事では、ナローウィンドウDIAデータセットをMascotで処理する具体例を紹介します。
Keywords: chimeric spectra, FDR