Mascot+Proteome Discoverer+ML 使用時に必要な設定変更 : MudPIT scoring無効化
Mascot Serverは、Thermo Proteome Discoverer™ (PD) などのソフトウェアパッケージと組み合わせて使用される事も多いです。最新のアップデート ver.3.1において、Mascotのinstrument設定内で機械学習アルゴリズムの内容を指定できる機能を追加しました。この件についてはASMS 2025でのポスターでも発表しています。初期の頃にご案内していた内容に、重要な注意事項が不足しておりました。この機能で機械学習を使用する際、MudPIT スコアリングを無効にする必要があります。この設定変更は、Protein Discoverer側がProtein FDR Validator ノードにおいて正しく計算するために必要な操作となります。
問題:なぜ同定タンパク質数が増えないのか?
この機能について紹介するASMSポスターでは、PRIDEプロジェクト PXD028735を使用しました。これは、Pyuveldeら(Scientific Data, 9(126), 2022)のA comprehensive LFQ benchmark dataset on modern day acquisition strategies in proteomics (Scientific Data, 9(126), 2022). のrawデータです。
Thermo Orbitrap QE HF-Xで測定した4回のtechnical replicatesデータであるサンプルalphaの条件AとBのrawファイルを解析に利用しましたが、実際の論文では3つの測定データのみが言及されているため、我々も条件を合わせてtechnical replicatesデータ1、2、3を使用しました。条件AとBは、ヒト、酵母、E. coli由来各タンパク質の混合比率が異なります。
Proteome Discoverer 3.2とMascot Server 3.1でファイルを処理した結果を見たところ、予想外のことが起こりました。MASCOTにてスペクトル予測(MS2PIP)とRT予測(DeepLC)を有効にした検索を行った際、Percolatorノードのみを使用した場合よりも多くの同定ペプチドが検出されたにも関わらず、タンパク質を同定数はむしろ減っているのです(以下の表)。
| No ML | Percolator node | Mascot ML | ||||
|---|---|---|---|---|---|---|
| Instrument | ESI-TRAP | ESI-TRAP | HCD2019:hela_lumos_2h_psms | |||
| #proteins | #peptides | #proteins | #peptides | #proteins | #peptides | |
| Identified | 4268 | 40964 | 6785 | 52935 | 6483 | 57098 |
| Quantified | 4241 | 33103 | 6442 | 40341 | 6298 | 42524 |
タンパク質数はPDの「Proteins」タブから、ペプチド数は「Peptide Groups」から取得し、いずれもデフォルトの1% FDRを適用しています。最初のケース(No ML)は機械学習を一切使用していません。2番目のケース(Percolatorノード)は標準のMascot検索を行い、その後Proteome DiscovererのPercolatorノードを実行します。最後の3番目のケース(Mascot ML)は、MascotにてMS2RescoreとPercolatorを使用して結果をrefinementし、その結果をPDに送っています。
3番目のMS2PIPとDeepLC(HCD2019:hela_lumos_2h_psms)を有効にしたケースにおいては2番目のPercolatorノードに比べて8%多くのペプチドが検出されているのに、同定タンパク質が増えるどころか減っています。これについて、何が起きているのか確認をしました。
解決策:MudPIT スコアリングを無効にする
解決策は予想外のものでした。Proteome Discoerer において、[Administration] -> [Processing Settings] -> [Mascot] -> [Mascot Protein Score] へ移動し、MudPIT スコアリングを無効にする設定を適用する必要があります:
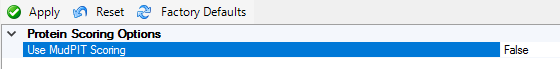
“study”を閉じて再度開き、consensus workflowを再実行します。ワークフロー内のパラメーターは変更する必要はありません。
処理が完了すると、ペプチドグループの数自体は変わりませんが、PDはより多くの同定タンパク質を識別できるようになります。
| No ML | Percolator node | Mascot ML | ||||
|---|---|---|---|---|---|---|
| Instrument | ESI-TRAP | ESI-TRAP | HCD2019:hela_lumos_2h_psms | |||
| #proteins | #peptides | #proteins | #peptides | #proteins | #peptides | |
| Identified | 4268 | 40964 | 6785 | 52935 | 6899 | 57098 |
| Quantified | 4241 | 33103 | 6442 | 40341 | 6566 | 42524 |
8%多い同定ペプチド、124の追加タンパク質(定量解析)が得られました。
この操作の意味について
理由はProtein FDR Validatorノードの仕様にあります。このノードは、タンパク質FDRを推定するためにタンパク質スコアまたはSum PEP Scoresメトリクスのいずれかを使用します。
Percolatorノード: PercolatorノードがMascotの結果を処理する際、Sum PEP Scoresを計算します。これは式で表すと -∑ilog10(PEPbest peptide, i)であり、各同定ペプチドに対して最も良い(最も値が小さい)PEPが選択されます。Protein FDR Validatorノードは、ターゲットタンパク質とデコイタンパク質間のスコア閾値を設定する際、Sum PEP Scoresを使用します。
Mascot ML: Mascotが結果をrefinementした際、-10log10(PEP)をペプチドスコアとして返します。ただし、Mascotノードは現在、ペプチドスコアからPEPを「展開」することはなく、これより下流のPDノードでは計算されたPEPを認識しません。その代わり、Protein FDR Validatorノードは、ターゲットタンパク質とデコイタンパク質間のスコア閾値を設定する際、タンパク質スコアを使用します。
Protein FDR Validator (Standard score使用時): MudPIT スコアリングが無効化されている場合、standard protein scoreが使用されます。Standard protein scoreは、タンパク質にアサインされた同定ペプチドのスコアの合計であり、各同定されたペプチドに対して最も良いスコア(最も小さな PEP)が選択されます。式で表すと、-10∑ilog10(PEPbest peptide, i)となり、Sum PEP Scoresと同じです。Protein FDR ValidatorノードがStandard protein scoreを使用する事で、同定基準値の設定はSum PEP Scoresと等しくなります。(係数10は無関係です。)
Protein FDR Validator (MudPIT score使用時):MudPITスコアリングは、確率的なアイデアとは異なる同定タンパク質順位付けとして設計されました。ペプチドのPSMスコアの使い方、スコア閾値の計算は複雑な式であり、Sum PEP Scoresとは同等ではありません。Protein FDR ValidatorがMudPITスコアを使用すると、ターゲットタンパク質とデコイタンパク質の間で適切な閾値設定を行うことができないのです。
理屈としては理解できるとして、ユーザー側から見て数値が適切かどうかをどう判断すればよいでしょうか?Proteome Discovererの結果閲覧において、Results Statisticsタブに移動し、Sum PEP ScoresまたはProtein Score行までスクロールダウンして内容を直接ご確認ください。これらの指標が手軽に判断できる材料となります。
| Minimum | Median | Maximum | Sum | |
|---|---|---|---|---|
| Sum PEP Score | 0.89 | 13.45 | 712.59 | 1,004,382 |
| Standard score (-10log10(PEP)) |
15.01 | 150.69 | 7274.10 | 10,702,554 |
数値の増減が指数関数的です。(なお数値が一致するのは、Mascot ServerとPDがPercolatorのcore features含め完全に同一で使用した場合のみです。)
新たに推奨するProteome Discovererの設定手順
設定手順を以下のように更新しました Using machine learning with Mascot Server 3.1 and Proteome Discoverer (5 pages, 291 kB)
以下簡単に手順をご紹介します。
ステップ1 : Mascot configエディターでMS2PIP:HCD2021のようなInstrument設定を作成し、refinement機能を有効にし、機械学習のパラメータを定義したmodel(トレーニングデータセット由来の名称)を選択します。
ステップ2:Mascotノード設定でMax MGF Sizeを増やします(既に設定済みの場合は不要です)。
ステップ3: MudPITスコアリングを無効にします(既に設定済みの場合は不要です)。
ステップ4:以下のように処理ワークフローを実行します:
![Mascot processing workflow in Proteome Discoverer: [Spectrum Files] to [Spectrum Selector] to [Mascot] to [Target Decoy PSM Validator]](https://www.matrixscience.com/images/Mascot_PD_processing_workflow_TD_validator.png)
ノード設定で、新しく作成したInstrumentを選択してください:
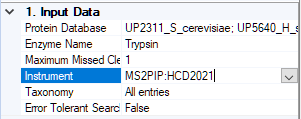
コンセンサスワークフローは、標準的なコンセンサスワークフローのうち適切なものを選択してください。いずれのケースにおいてもProtein FDR Validatorノードを使用してください。
Keywords: machine learning, MS2PIP, Percolator, Proteome Discoverer