BLIBスペクトルライブラリをMSP形式に変換(およびさらなる逆変換)してMascot Serverで使用する
Mascot ServerはNIST MSP形式のスペクトルライブラリを作成・検索できます。しかし、Skylineソフトウェアの一部であるBiblioSpecが生成するBLIB形式のスペクトルライブラリは使用できません。私たちはBLIBからMSPファイルへ、または逆方向にMSPからBLIBファイルへ変換可能なスクリプトを開発しました。この変換スクリプトを使う事でCarafeや類似の予測ソフトウェアで生成されたin silicoライブラリをMASCOT Serverで検索できるようになります。SkylinedはNIST (.MSP) ファイルを読み込む事ができますが、MSPライブラリをBLIB形式に変換することで、他のスペクトルライブラリ(SL)検索エンジンでの利用が可能となり、ソースに関わらず複数のプラットフォームで一貫して同一ライブラリを使用できるようになります。
今回、テスト目的で、マイコバクテリアデータセットのBLIBファイルを変換しました。ライブラリのサイズは50メガバイトから2ギガバイトまで様々でした。また、ライブラリを一方の形式から他方の形式に変換し再び元に戻す往復テストも実施し、元のライブラリと再生成されたライブラリに差異がないことを確認しました。この変換プログラムは弊社のGitHubリポジトリからダウンロード可能です。
BLIBからMSPスペクトルライブラリへの変換プログラムのインストールと使用方法
BLIBからMSPスペクトルライブラリへの変換プログラムはPerlで書かれており、Mascot Parserライブラリを使用します。WindowsではStrawberry Perl 5.32.1.1を推奨します。Linuxでは通常、LinuxディストリビューションのレポジトリからPerlがインストールされます。Mascot ParserはUnimodによる修飾やMSPファイルの読み取りでルーチンとなっている処理を提供しています。Mascot Parserのライセンスでは、作成アプリケーションに対してライセンス料を請求しない限りライブラリを使用したコードやアプリケーションの開発者による配布が許可されています。商用ライセンスも利用可能です。
変換プログラムをダウンロードし、同梱のreadmeの説明に従ってMascot Parserをインストールしてください。完了後、コマンドライン上でBLIBとMSPフォーマット間の変換を簡単に行うことができます:
# BLIB からMSPへの変換 (拡張子から自動的に判別されます)
perl blib2msp.pl -i library.blib
または、
#MSPからBLIBへの変換 出力ファイル名をオプションにて指定可能
perl blib2msp.pl -i library.msp -o output.blib
修飾
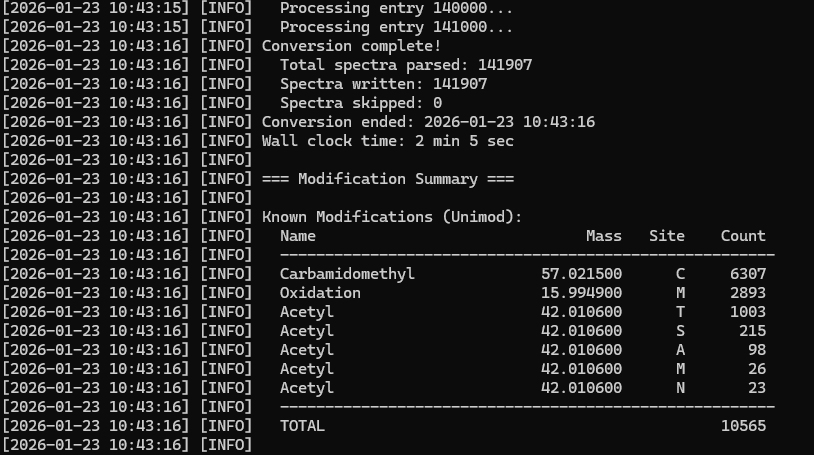
スペクトルライブラリ変換終了時、ライブラリ内の修飾に関する情報をまとめた表が報告されます:
FASTAデータベースなしで1つまたは複数のスペクトルライブラリを検索する場合、Mascotはスペクトルライブラリ内に含まれている修飾情報を自動的に使用します。検索フォームで固定/可変修飾を指定する必要はありません。
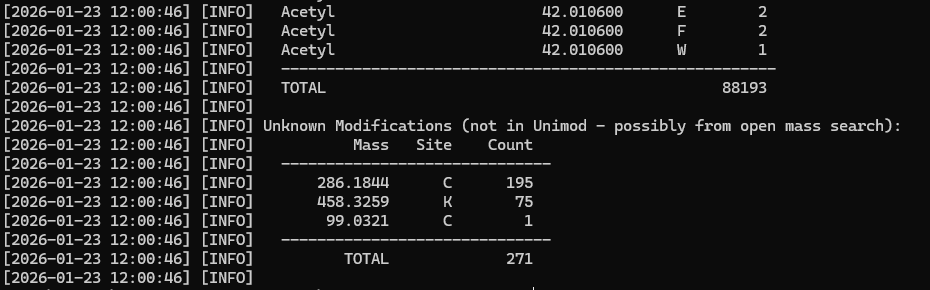
BLIB スペクトルライブラリがオープンマス検索から作成された場合やUnimodに含まれない修飾を使用していた場合、変換プログラムは単なる質量の数値として報告します。
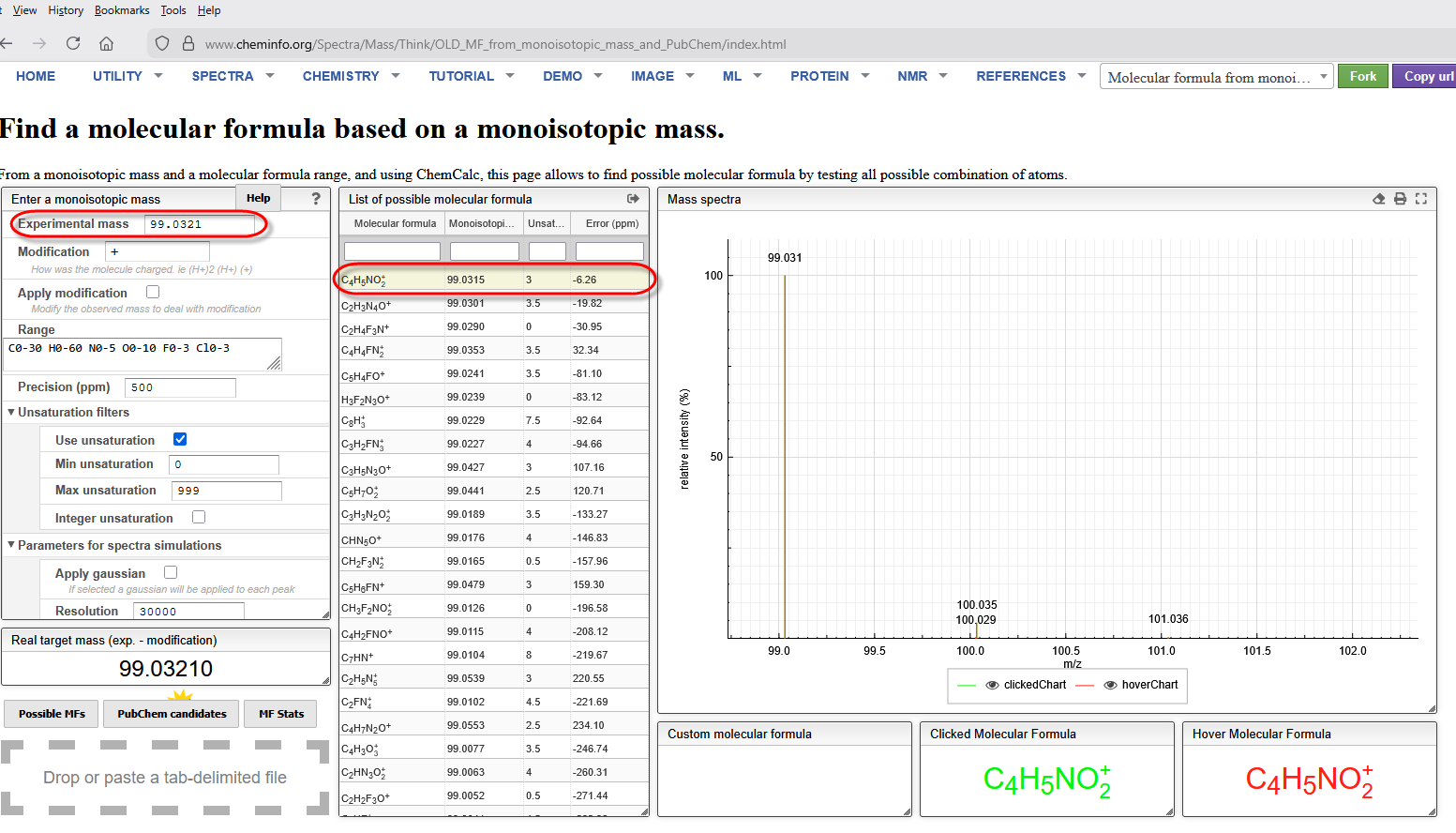
表示内容だけからでは質量差が何を意味しているかを知ることはできないですが、スペクトルライブラリを検索に利用できるようにするためにはこのままMascot Serverに追加する必要があります。今回の例では、最初の2つは質量から推測可能です:Cys上の+ 286.1844はTMT6plex + カルバミドメチル化に相当し、Lys上の458.3259はN末端と側鎖へのTMT6plex付与に該当します。このように、質量に対応する分子組成を推測し、Mascot Serverにカスタム修飾として追加する必要があります。今回の例でいえば、未知のCys上の+99.0321について、Cheminfo.orgのChemCalcツールで候補化学組成リストを作成し、質量誤差が最も近いものを選択して登録しました。
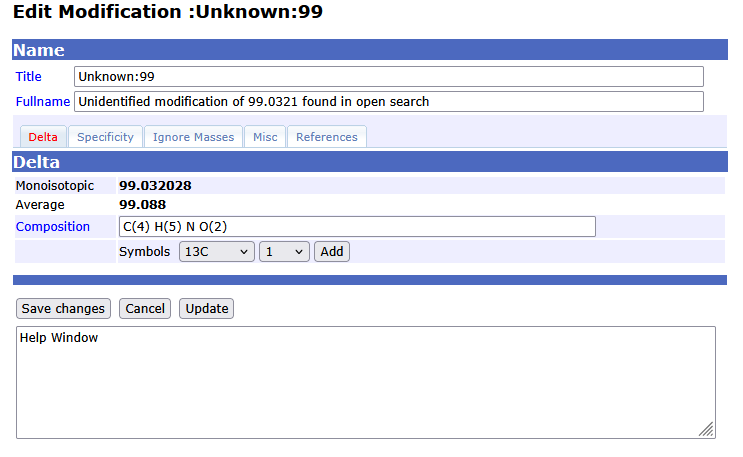
該当エントリを選択後、右クリック→エクスポート→選択項目を選択すると、クリップボードにコピー可能な化学式が表示されるポップアップウィンドウが開きます。ブラウザでMascot Server→Configuration editor→ Modificationsエディタを開き、新規修飾を作成後、化学式を組成欄に貼り付けます。組成の編集の際には、元素数を括弧で囲み入力してください。
Specificity タブで修飾部位のアミノ酸に関する情報を設定し、保存します。ここでは、オープン検索で検出された他の未知修飾と同じ命名形式(例:Unknown:99)を使用することを推奨します。99 flake ではないですよ。
BLIBライブラリは更新された新しいunimodファイルで変換されていないため、BLIBからMSPに変換する際、以下のいずれかの方法で対応してください。1:ローカルのMascot Serverディレクトリ(mascot\config\unimod.xml)のコピーを使用して、BLIBからMSPへの変換を再実行する。2:mascot\config\library_mod_aliasesに、以下のような質量値のエイリアスを追加する
"286.1844" = "TMT6plex + Carbamidomethyl" "458.325" = "Double TMT6plex" "99.0321" = "Unknown:99"
最終結果は同一ですが、私の好みは前者、ライブラリの再変換です。
生物種が混ざっているライブラリ
複数の生物種を含むサンプルやメタプロテオミクスから生成されたスペクトルライブラリに役立ついくつかの機能をご紹介します。例えば、BSAやその他のタンパク質を含む培地で培養された感染創傷の綿棒検体分析から生成されたメタプロテオミクスのスペクトルライブラリなどです。この検体には感染生物(ヒト/動物)、感染細菌、培養培地由来など様々なタンパク質が含まれます。
Mascot Serverでライブラリを使用可能にする際、スペクトルライブラリ定義で指定されたFASTAデータベースへのタンパク質とのマッピングも自動的に実行されます。あるいは、ライブラリ作成時に--fasta-dirというオプションを用いることも可能です。スペクトルライブラリを作成するレベルでこれを実行すると、マッピング過程で細菌・ヒト・BSAタンパク質を網羅する複数データベースを併用できるというメリットがあります。ただしマッピング実行には追加のPerlモジュールインストールが必要です(詳しくは関連readmeファイルに手順が記載されています)。種別マッピングを実施すると変換処理時間が長くなりますのでご注意ください。
この他にもメタプロテオミクス解析に有用なユーティリティースクリプトが2つ用意されています。1つはデータベース内のペプチド配列リストを生成するスクリプト、もう1つはスペクトルデータのみを含むようにデータベースをフィルタリングするスクリプトです。これらの使用方法については続編の記事で解説します。
Keywords: metaproteomics, spectral library