Daemonの新機能 Quantitation Summary で、発現解析データの集計をより簡単にする
定量プロテオミクスでは様々なサンプル・測定データを取り扱います。異なる細胞や実験処理由来のデータ、あるいは時系列での状態変化を見るための測定であったり、サンプルが分画されていたりする事もあります。 定量データの再現性チェックのため、繰り返し実験(Replicate)が行われる事もあります。これらの解析結果を組み合わせ、統計的手法を適用して意味のある情報を抽出し、グラフや表として報告するまでの過程は非常に複雑な作業です。
定量データを駆使してデータをまとめる具体的な手段として、Rなどの言語によるスクリプトを利用したり、 Perseus(Max Planck Institute)などのソフトウェア利用などが挙げられます。マトリックスサイエンス社日本法人の取り扱いソフトウェアでも、Scaffold Q+やQ+Sがこれらの解析に対応しています。これらのソフトウェアを利用する時には基本的にスプレッドシート形式の入力データが必要で、行はタンパク質に対応し、列には様々なサンプルの発現データの定量値または定量値の比の形で含まれたデータとなります。
Mascot Daemon 2.7 の新機能として、このようなスプレッドシート形式に対応するデータ出力が可能となりました。Daemon上で複雑な統計解析などはできませんが、別ソフトウェア上で解析を行う際に便利なデータの属性付与をDaemon上で行う事ができます。ラベルフリー法や、isotopicなラベル法(SILACなど)、レポーターイオンによる定量法 (iTRAQ,TMTなど)などに対応しています。この記事では、Daemonで如何にして上記のようなスプレッドシートのデータを出力するかについて説明しています。またこの次の記事では出力したファイルを使ってレポートやチャートを作成する例をご紹介予定です。なお、今回並びに次のブログ記事に関連する発表資料が、こちら(PDF、ビデオ)にもございます。
MASCOT Daemonで行った定量解析結果を、スプレッドシート形式のテキストである Quantitation Summaryとして利用するためには、ユーザーの操作で各検索結果にアノテーションをつける必要があります。この、定量解析からアノテーションをつけていく作業の流れについて、具体例を使って説明します。例ではデータレポジトリサイト PRIDE に保存されているデータPXD001385から、12のrawファイルをダウンロードして利用しています。スパイクされた大腸菌のng量を表すサンプル名称 3, 7.5, 10, 15 という4種類のサンプルについて、3回の繰り返し実験を行った計12回の測定です。(15ngサンプルと比較して、5倍、2倍、1.5倍の変化をシミュレートしたものです。)
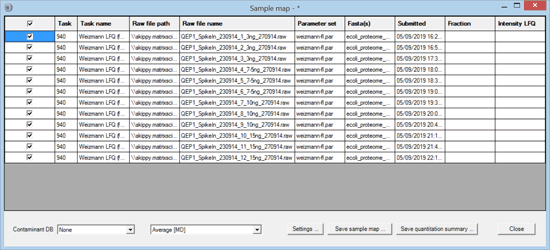
検索にはピーク抽出と定量解析にMascot Distillerを使用して、Daemon の1タスクで12サンプルを処理しました。Distillerのピーク抽出パラメータとして Thermo Q Exactiveデータ向けの典型的な設定を使用し、MASCOT検索における対象データベースとしてはヒトおよび大腸菌プロテオームとコンタミ検証用データベースを使用しています。また定量解析のパラメータとして、ラベルフリーの定量法の1つであるAverage [MD]をMASCOTで選択しています。検索終了後、Daemon上で該当タスクを選択し右クリック→Quantitation Summary → New sample map と選択します。すると以下のようなサンプルマップテーブル(ダイアログ)が現れます。
サンプルマップテーブルでは、1つまたは複数のタスクから選択された各結果ファイルに対して識別/データ集積のためのラベル付けを行います。定量解析では大量のデータ解析が必要になる事が多々あります。例えば3つのコントロールのサンプルと何かしらの処理がされた3つのサンプル、計6つの生物学的サンプルにおけるラベルフリー定量解析において、さらにそれぞれのサンプルが4つの時系列、6つの分画、それらが3回繰り返し実験をされているとすると、6 x 4 x 6 x 3 = 432 の解析データとなります。「あるタンパク質について、コントロールと何かしらの処理がされたサンプル間の差は有意に差があるか?(繰り返し実験のデータも利用して)」といった問いに答える解析を行うためには、これら大量の定量解析データに対して識別や繰り返し単位のラベル付けを正しく行い、適切に処理する必要があります。
Daemonで作成したサンプルマップテーブルは、識別や繰り返し単位のラベル付けをできるだけ効率的に行うために様々な項目で並び替えをすることができます。今回の例ではrawファイル名で並び替えをするだけで十分です(先頭行の項目名のところをクリック)。データ数がもっと多い複雑なデータの場合(分画が複数に分かれていてデータ数が非常に多くなっている場合など)は、他の項目であるファイルパス、タスク名、検索時間などの並び替えも駆使するとよいかもしれません。
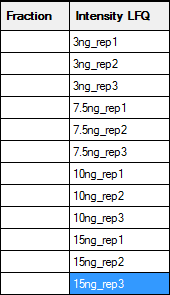
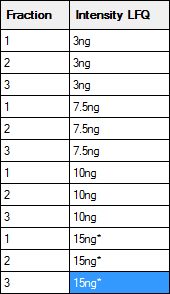
"fraction"とかかれた列は、fraction番号などを記入してもらう意図で準備をしていますが、必ずしもこの用途で使っていただく必要はありません。必要なければ空欄のままでもいいですし(下図 左側)、例えば今回のような例で繰り返し実験への割り振り番号として 1~3を記入しても良いでしょう(下図 右側)。入力した属性情報は用途に応じて様々な記述が可能です。
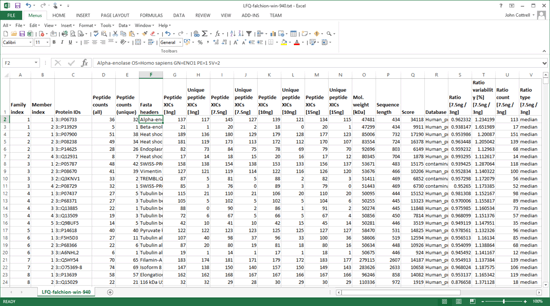
作成された サンプルマップはファイルとして保存することもできますし、作成途中でセーブして後でロードし、少し内容を変えてから再利用することもできます。「Save quantitation summary ...」を選択すると、データに関していくつかの整合性チェックが実行されます。問題ないと判断された後、Quantitation Summary が作成されます。出力されたファイルはテキストファイルで、スプレッドシートを開くことができるEXCELなどのアプリケーションでも、テキストエディタでも開くことができます。
DaemonにおけるQuantitation Summaryでは、MASCOT ServerのProtein Family Summary report (query数が多い時にデフォルトで表示されるMASCOTの検索結果画面)と同じ基準でタンパク質のグループ化が行われます。データベース検索の解析結果で得られた同定タンパク質のリストと、定量解析の際に行うリストの内容が厳密に一致し、その後の解析における問題が生じません。詳細はMASCOT DaemonのHELPをご覧ください(ソフトウェアから開きます)。
次のブログでは、レポートやチャートを作成するためにサマリーを使用する例を紹介します。
Keywords: export, Mascot Daemon, Mascot Distiller, quantitation, statistics, tutorial