Daemonの新機能 Quantitation Summary : 出力データを使った統計解析の実施やグラフの作成例
先月の記事ではMascot Daemonで 属性情報を付与し集計し直した定量情報のスプレッドシート: Quantitation Summaryを作成する方法について説明いたしました。各行がタンパク質に対応し、列は発現データの定量値または定量値の比の形で含まれたデータとなります。
Quantitation Summaryとして出力したファイルは、スプレッドシートを開くことのできるEXCELのような基本プログラムでもファイルを読み込みデータ処理を行うことができます。しかし基本的な表計算ソフトウェアを使って複雑な統計解析を行うためのデータ処理をするのは一苦労です。より効率的である、専用のソフトウェアを使った解析をお勧めいたします。この分野でよく使われているソフトウェアは、Max Planck研究所が開発したソフトウェアPerseusです。グラフィカルなユーザーインターフェースを使ってデータを取り扱う事ができます。スクリプトを使ったプログラミングでデータ処理をしたいという場合は、'R'の使用をお勧めします。膨大な種類の統計ツール、グラフ表示ツールを使用することができます。Bioconductorはゲノムやプロテオミクス解析用のアプリケーションを集めたパッケージです。 ver.3.11 の段階で、プロテオミクス用として135、質量分析装置のデータ解析用に91のパッケージが準備されています。
ここでは、Bioconductorの中のパッケージの1つであるDEP(Differential Enrichment analysis of Proteomics data) という名称のパッケージを使用して数行のスクリプトで実現できる分析をご紹介します。
今回のご紹介で利用する解析データは、非小細胞肺がんにおける発がん性マイクロRNAを同定するための研究データです。
microRNAs with AAGUGC seed motif constitute an integral part of an oncogenic signaling network
Y Zhou, O Frings, R M Branca, J Boekel, C le Sage, E Fredlund, R Agami & L M Orre
Oncogene volume 36, pages731–745(2017)
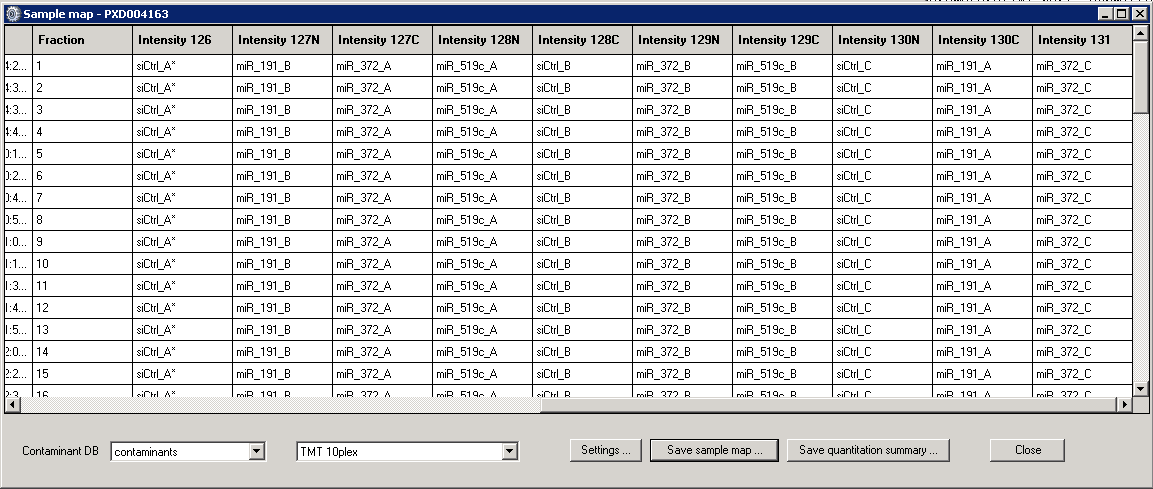
定量解析手法は10 plex TMT です。PRIDE上のデータPXD 004163から、72のデータをダウンロードしました。解析対象の10ピークについては、コントロールに3つ、1種類のmicroRNAのデータには3つ、他の2種類のデータに対しては2つずつ対応させています(3+3+2+2=10)。peptide FDR 1% を同定ペプチドの基準としています。
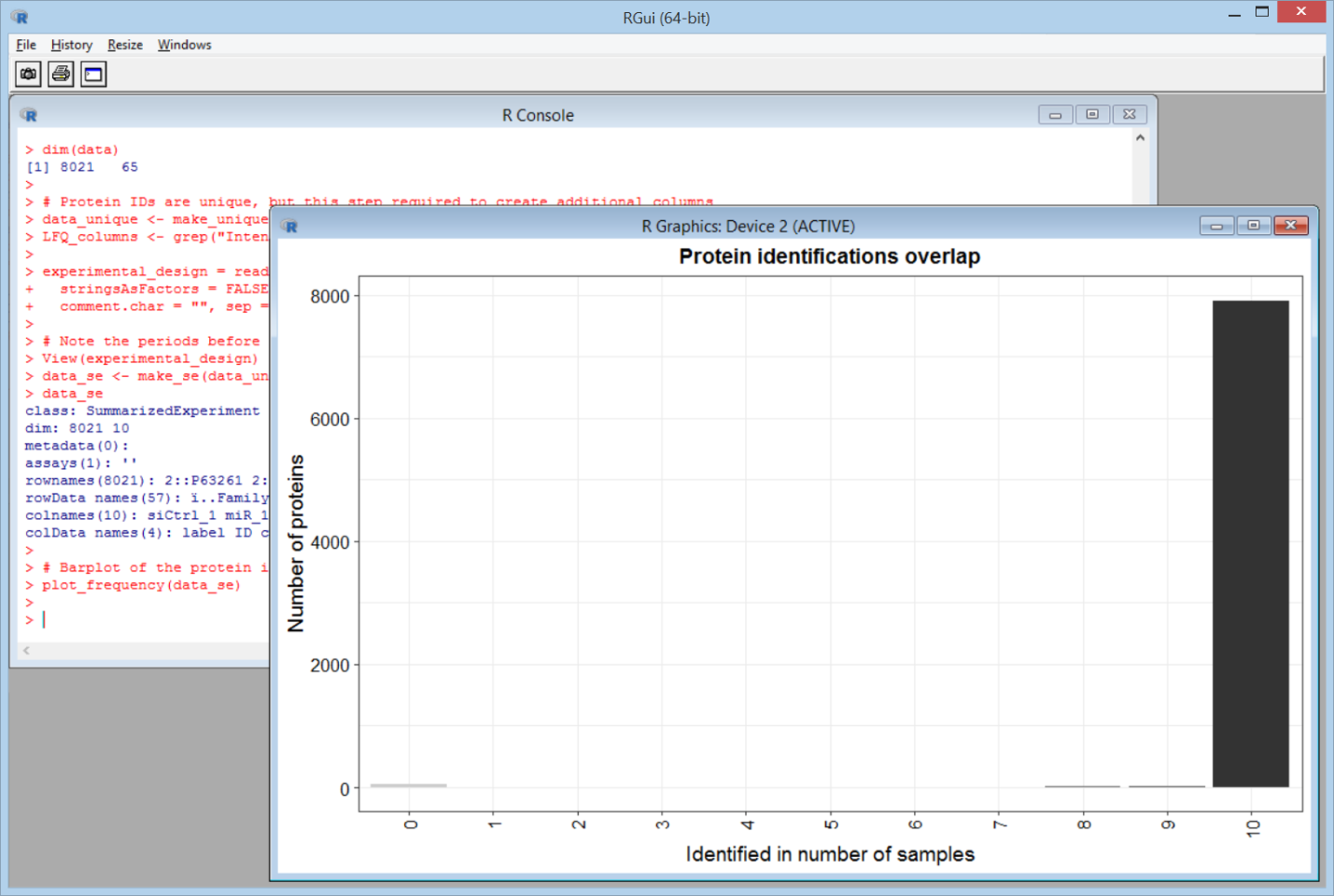
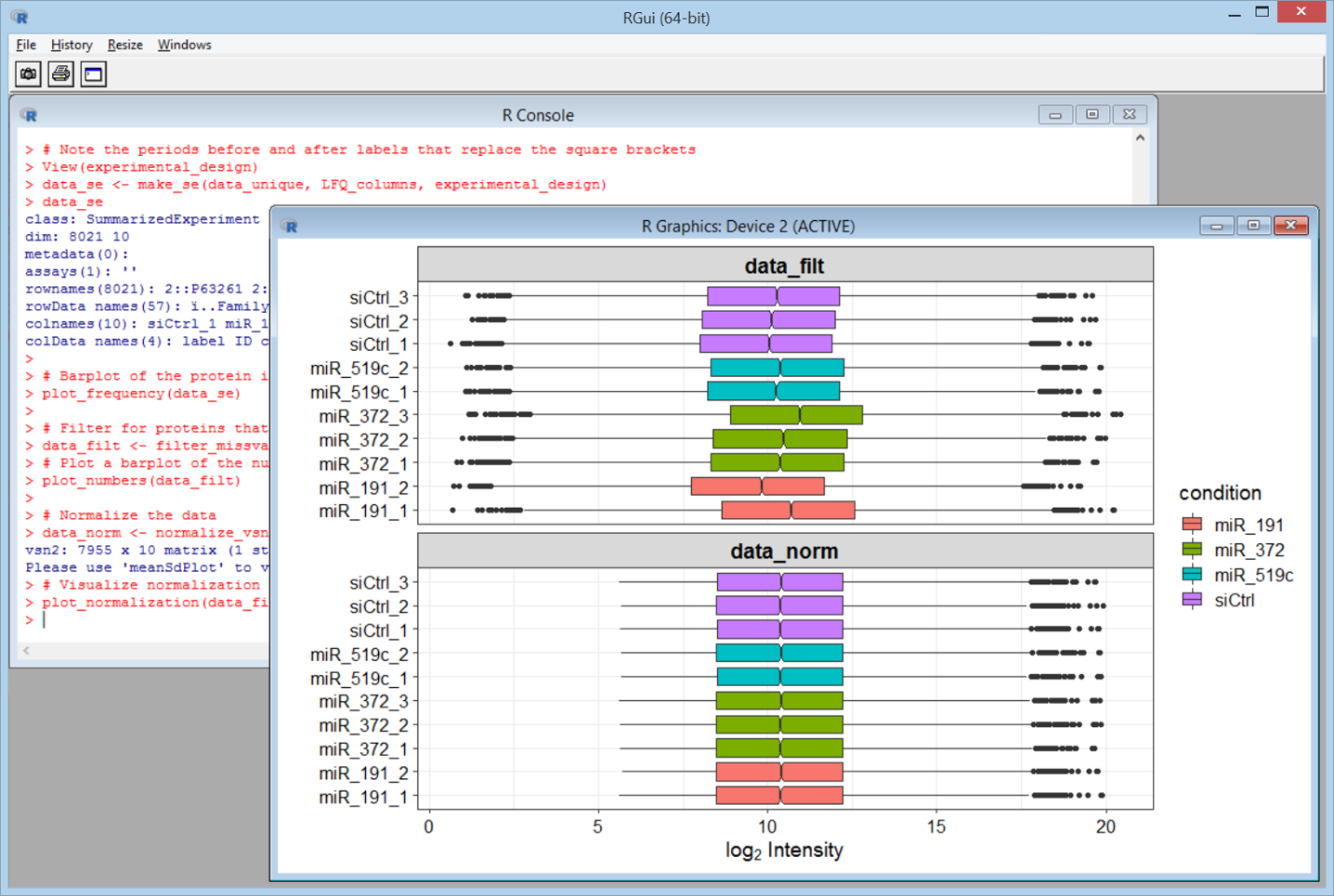
DEPパッケージを使用するとグラフを簡単に作成することができます。一例としてまずはデータの品質管理確認のための棒グラフをご紹介します。各タンパクにおいて10の定量解析用ピークのうちはっきりピークの値がでている数をカウントしこれを横軸に、そして縦軸にはタンパク質数を配置した棒グラフです。8025個のタンパク質のデータ解析で、使用されている 10チャンネルのほとんどでmissing value が少ない、良いデータであることがわかります。
続いて、normalization 前後の intensityの値の分布について、box plotとして表現された図です。
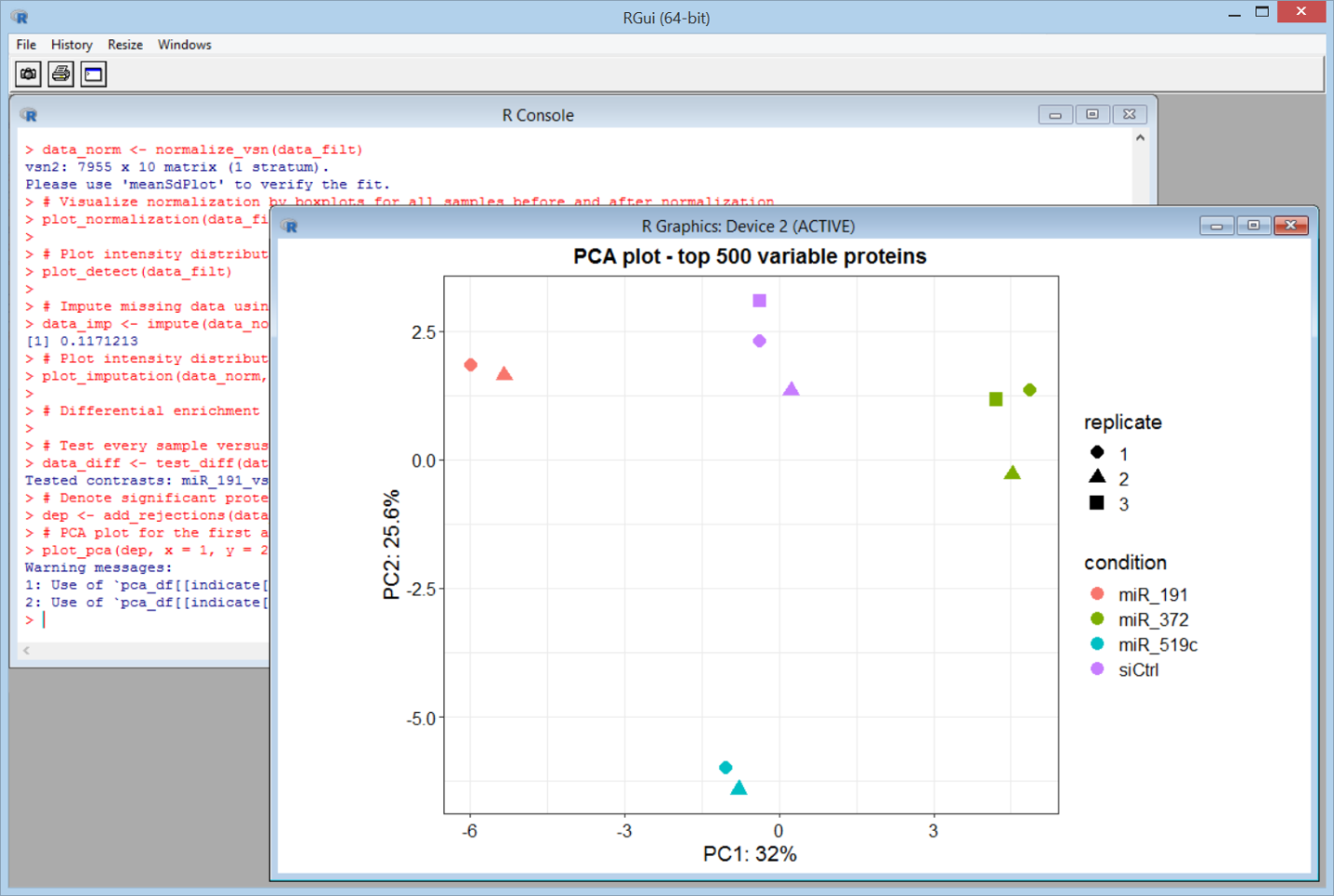
PCA解析も行うことができます。サンプル別にきれいにデータが分かれていることが確認できます。
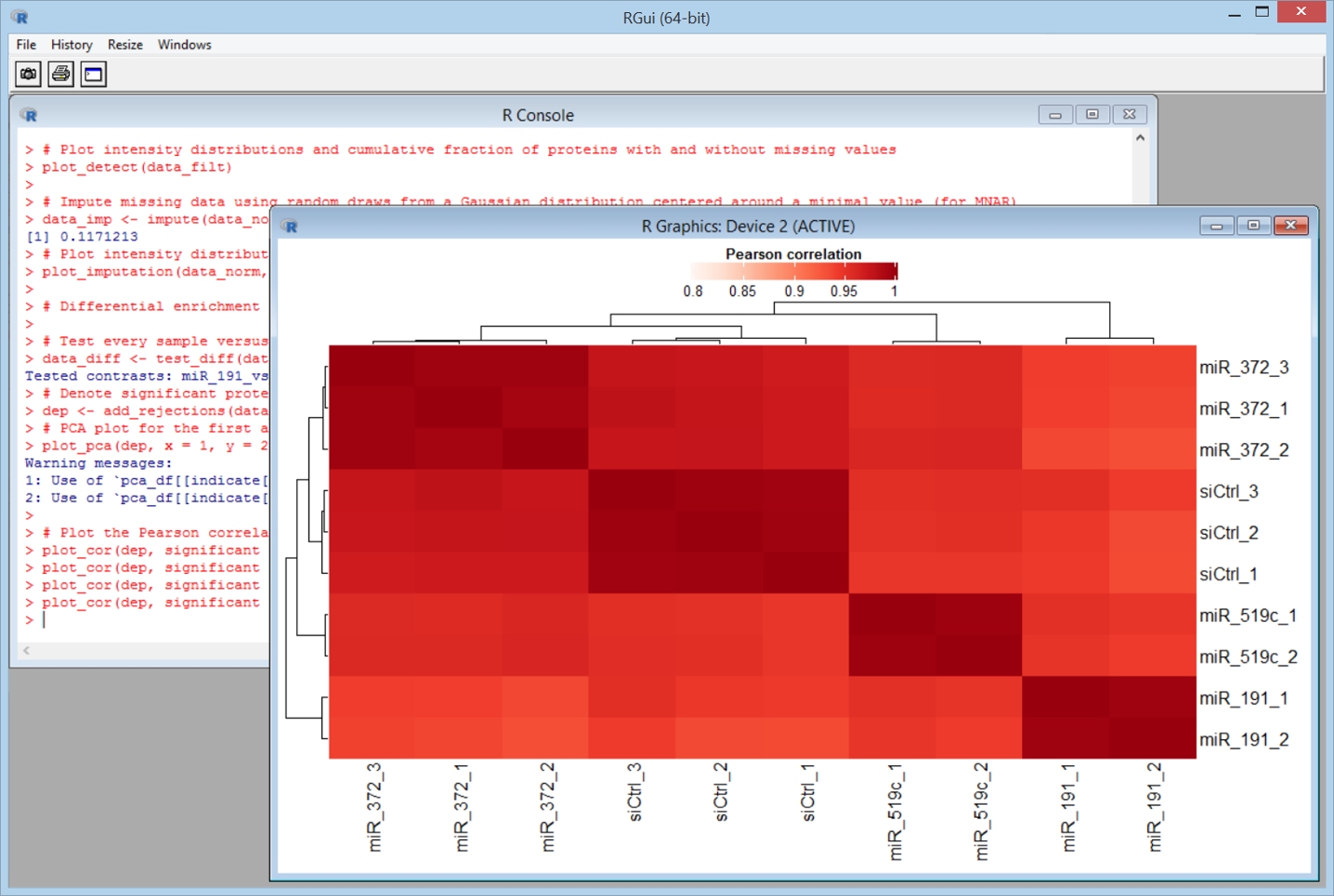
データのクラスタリングのひとつ、ヒートマップ解析です。
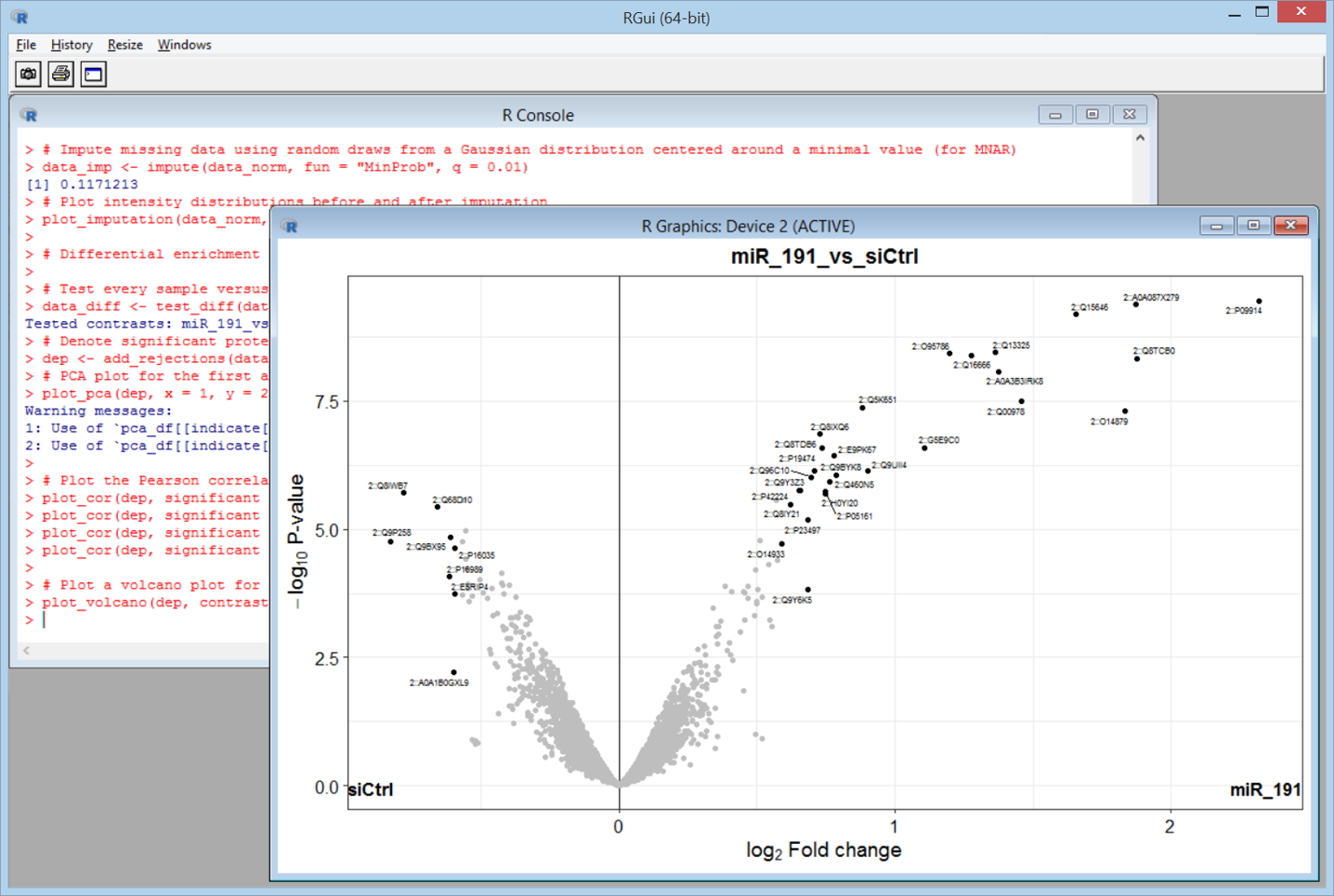
以下の図はvolcano plot です。2サンプル間の発現の差をチェックしています。横軸の左右へ行くほど2サンプル間の値の比が大きく、縦軸の上に行くほど検定のp値が小さくます。比だけでなく検定結果も加わっており、繰り返し実験の分散状況も加味した変動の検証を行うことができます。
Sample map や Quantitation Summary に関するより詳しい情報についてはDaemonのソフトウェア内のHELPページをご覧ください。また今回紹介したBioconductor のパッケージは、ほとんどがその機能について文書化されており、ウェブ上にも多くの R のチュートリアル資料があります。
作成された サンプルマップはファイルとして保存することもできますし、作成途中でセーブして後でロードし、少し内容を変えてから再利用することもできます。「Save quantitation summary ...」を選択すると、データに関していくつかの整合性チェックが実行されます。問題ないと判断された後、Quantitation Summary が作成されます。出力されたファイルはテキストファイルで、スプレッドシートを開くことができるEXCELなどのアプリケーションでも、テキストエディタでも開くことができます。
このページでご紹介したグラフの Quntitation SummaryファイルとRのコマンドはここから解析に使用したファイルを取得してご確認いただく事ができます。
訳者 補足
Rを使った定量プロテオミクスに未経験ながらもご興味がある方は是非、今回解析に使用したファイルをダウンロードしてファイルの中をご覧ください。スクリプトの記述を見て試しに動かしていただく事で、より Rの使い方がイメージしやすくなるのではないかと思います。
- PXD004163.txt ...Quantitation Summary Report, Daemonが出力したスプレッドシートです
- PXD004163_DEP_R_script.txt ... Rのスクリプトです。テストで実行される際は中身を開き、読み込むファイルの置き場所を書き換えてご利用ください。
- PXD004163_exp_des.txt ... Quantitation Summary Reportで定義しているラベル情報から、Bioconductorの計算で利用する「Condition」と「Replicate」項目の定義をしているファイルです。
詳細は検索などをしていただければと思いますが、以下、最短で動かすところまでの操作例をご案内いたします。
- ファイルをダウンロードし、特定フォルダに設置
- Rをインストール
- Rを起動
- R上からBioconductorをインストール
- 作業場所の設定 :setwd ("current path")や、「ファイル」→「ディレクトリの変更」など
- DEPパッケージの読み込み : require (DEP)
- メニューの"新しいスクリプト" → PXD004163_DEP_R_script.txt の内容を貼り付け、1行ずつ上から実施
Keywords: export, Mascot Daemon, Mascot Distiller, quantitation, statistics, tutorial