データを分けて打ち勝つ。Mascot Distiller 2.8.2と分画ラベルフリー定量分析
Matrix Science ではMascot Distiller の最新版、2.8.2をリリースいたしました。 今回の新機能では分画されたサンプルのラベルフリー定量をサポートしたことが大きな特徴となります。
Distillerが分画データを扱う時には、プロジェクト内の個々のRawファイルの内容をラフに並べて結合して一つの大きな仮想TICを作成します。あるサンプルファイルでペプチドマッチが見つかり、他のサンプルファイルでは見つからなかった場合、その仮想TICのアライメントを使用して、一致しなかったファイルでのペプチドXIC検出の開始点を計算します。 これについては、以前のブログ記事(日本語版、英語版)で詳しくご紹介しています。
問題は、生成されたコンセンサスはすべての分画の単純な集合体であることです。データをつなぎ合わせる根拠となるようなペプチドの重複がほとんどないので、正確な表現にならないという事です。
Mascot Distiller 2.8.2では複数のコンセンサスの作成されるよう仕様を変更しました。この方法を採用する事により、分画されたサンプルを適切にサポート可能となっただけでなく、プログラムがコンセンサスが見つかった対応データからだけ相手のコンセンサスを探す事が可能となり、すべてのファイルからペプチドを探す時間の浪費を避け定量計算が高速化されました。
GelC-MSの例
分画されたLFQ実験の最も一般的な例として、'GelC-MS' アプローチがあります。 タンパク質サンプルは、まず1次元の電気泳動で分離し、ゲル上のレーンを一定数の等しいサイズにスライスと消化酵素処理を行い、消化物に対してLC-MS/MSを実行します。 このゲルスライスが事実上、サンプルの別々のフラクション単位にあたります。
Distiller 2.8.2での分画データに対するサポートを実証するため、PRIDEリポジトリで公開されているデータセット、PXD029062からデータを取得しました。HeLaとHeLa KO30細胞の2つのサンプルで構成されます。 各レーンから4つのスライスを取ったため、合計8つのrawファイルが得られました。
このrawファイルをMascot DaemonとMASCOT Distiller(+Quantitation Toolbox)を使って検索を行いました。 Mascot Distiller 2.8.2でピーク抽出処理を行い、生成されたピークリストファイルをMascot Server 2.8.1で、データベースUniProt Human reference proteomeに対して検索を行いました。定量計算のパラメータとして、今回の目的にカスタマイズしたLabel-free Quantitation Methodを使用しました。
Daemonによって生成された.rovプロジェクトファイルを元に、Mascot Distiller GUIでmultifileプロジェクトを作成し、新機能の効果を確認するため2種類の方法で定量計算をしました。
- Distiller 2.8.2の新しい設定を使用して、一致する分画だけでアライメントを実施した計算
- Distiller 2.8または2.8.1で得られるのと同じ結果となるよう、すべてのファイルを対象としたアライメントを実施
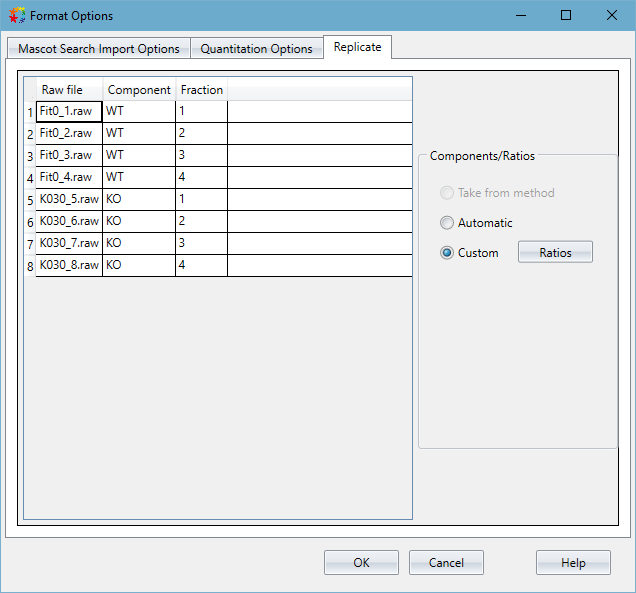
下の図1は、Distiller 2.8.2におけるコンポーネントと分画の設定画面です。コンポーネントと分画の組み合わせの単位でユニークであればよく、サンプル名が同じでも異なる分画番号が与えられていれば良いので、1つのコンポーネントに複数のRAWファイルを関連付けることができるようになりました。分画番号を手入力するのは面倒ですが、代わりにセルの範囲を選択し、右クリック→「Addinteger series」をクリックすることで自動的に記入する事もできます。
Click to view full size image
図1:プロジェクトのRawファイルに対してコンポーネントと分画を記入する設定画面。最初の列にプロジェクトのRAWファイルが、2,3列目はユーザーが編集可能な "Component "と "Fraction "の情報を記入する表です。 ComponentとはサンプルIDの事で、このデータセットではW.T.とK.O.の2つのサンプルがあり、図のようにファイルの「Component」列のセルにその情報を入力しています。 もし1つのサンプルに複数のtechnical replicates,あるいはbiological replicatesがある場合は、その情報をコンポーネントの名称に反映させておいてください。
すべてのファイルを結合せず独立したデータとして互いにアラインメントしたい場合、「Fraction」カラムを空白にしてください。その場合、コンポーネントだけでユニークな名称となるようにする必要があります。
表1は、全ファイルをアライメントした場合と、一致する分画のみをアライメントした場合の結果を示しています。 どちらの場合も、合計1417個のペプチドが定量されました。 一方かかった時間はすべての分画データをアライメントした場合26分かかるのに対し、一致する分画のみをアライメントした場合11分でした。これは、例えば分画3でのみ同定されたペプチドを探すのにDistillerが分画1、2、4のファイルから探すという無駄な時間を費やさないためです。
さらにこの仕組みを導入した効果により、個々の分画をアライメントした場合1137の定量化されたペプチドが質の高いデータとみなされる一方、全ての分画データをアライメントした場合は569に留まります。品質の面でも良い結果を得ることができました。
| Align Everything | Align Fractions | |
|---|---|---|
| No. peptide ratios | 1417 | 1417 |
| Time taken (min) | 26 | 11 |
| No. valid peptide ratios | 569 | 1137 |
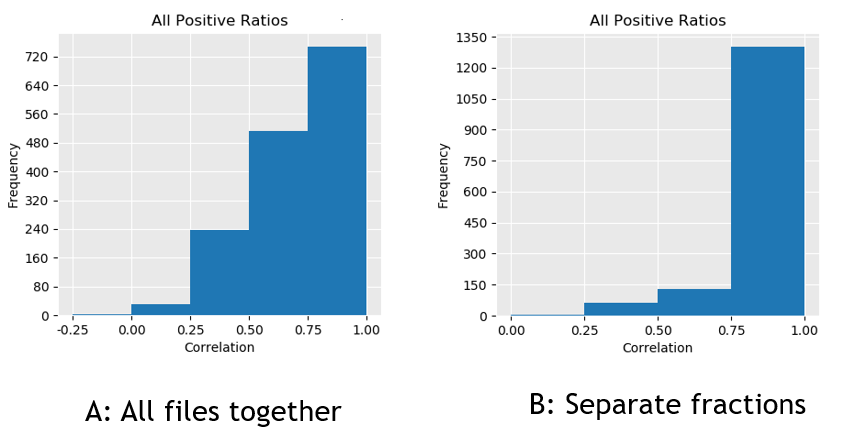
Mascot Distillerで使用している、データの品質評価の基準値は、入力データ側と理論値側のプリカーサー同位体分布の相関係数です。もしDistillerに全てのデータを対象としたアラインメントを実行させXICを探させた場合、データに多くのジャンク・ノイズを追加してしまい全体の相関を低下させてしまいます。一方一致する分画でのみアライメントした場合では相関が0.75から1.0の間のペプチドの数が、~720から~1300に増加しています(図2) 相関係数のデフォルトの閾値は0.8なので、対応する分画だけでアライメントする事でより多くの有効な定量データが得られます。
Click to view full size image
図2:実測値と理論値のプリカーサー同位体分布の相関係数の分布について、(A)全ファイルをアライメントした場合と(B) 対応する分画ファイルのみでアライメントした場合の比較。
Mascot Distillerのライセンスをお持ちの方は、無料で2.8.2へのアップデート可能です。Mascot Distillerのライセンスをお持ちでない方は、30日間の試用が可能です。詳しくは弊社までご連絡ください。
Keywords: label-free, Mascot Distiller, quantitation, replicate