Mascot Error Tolerant 検索で、狭いウィンドウ幅のDIAデータ解析から想定外の修飾を見つける
Mascotによるスペクトル中心のDIA解析に対するアプローチでは、ペプチド中心のアプローチと比べて可変修飾、切断ミス許容数、タンパク質配列に関する制約がありません。今月の記事では、Mascot Error Tolerant検索を使用する事で、狭いウィンドウ幅のDIAデータから想定外の可変修飾を同定する解析についてご紹介します。
MS/MSスペクトルと有意なアミノ酸配列の一致が得られない場合、多くの理由が考えられます。その中でもよくある理由は以下の3つです:
- 酵素の非特異性
- 想定外の化学修飾や翻訳後修飾
- ペプチド配列がデータベースにない
MASCOTのError Tolerant 検索は、化学修飾や翻訳後修飾の包括的なリストと置換残基マトリックスを使いながら検索したり、酵素特異性を緩和した検索を行ったりする事で、これらの問題に対処します。
Error Tolerant検索は2段階のプロセスです。 第1段階では、指定された検索パラメーターを使用して標準的な検索が実行されます。第2段階での検索では検索対象のタンパク質を、第1段階での検索において1つ以上の有意なペプチドがアサインされたものだけに絞り込み、代わりに検索空間を広げた第2段階の検索を行います。2段階目の検索が完了すると、両方の検索結果を組み合わせまとめたレポートが生成されます。 Error tolerant検索の1段階目の検索ではパラメーターの設定にいくつかの制約があります。その点についての詳細はヘルプで説明されていますのでご興味がある方はご覧ください。
前回のブログ記事(英語版、日本語版)で使用したPRIDEデータセットから前立腺がん患者のrawファイルの1つ(P1.raw)を取り出し、Error Tolerant検索を実施しました。 スペクトルはDIAモード、8m/zの分離ウィンドウで取得したデータです。 1段階目の検索で使用した検索設定は前回と同じとしました。MascotのError Tolerant 検索向けの統計モデルを適用して、マッチング内容の質と同定基準値の評価を行います。同定基準の目安としてPSM FDR1%という基準が、1段階目の通常検索並びに2段階目のErrort Tolerant検索の両方に適用されました。同定基準を超えたPSM数とペプチド数について、1段階目、並びに1段階目と2段階目を併せた検索結果での数が以下の表1です:
| No. significant PSMs | No. significant sequences | |
|---|---|---|
| First pass | 37231 | 5829 |
| Combined first pass + ET | 44066 | 6901 |
ご覧のように、Error Tolerant検索を適用する事により、6835 個の有意な PSM と 1072 個のペプチド配列が追加で見つかっています (1% PSM FDR)。Error Tolerant検索を実施したMascotの結果レポートには、2段階の検索を結合した結果におけるペプチド修飾の内訳が表示されます。
以下、Error Tolerant検索を実施する事でリストアップされたペプチドのいくつかを詳しく見ていきます。
非特異的切断
このデータセットで報告されたError Tolerant検索でのトップスコアは "非特異的切断(Non-specific cleavage) "でした。非特異的切断ペプチドの総数は1908でした。Error Tolerant検索では検索空間が拡張され、選択された酵素が半特異的、すなわちペプチドの一端のみが開裂特異性に一致し、もう一か所は任意の場所で切断します。それに加え切断ミス許容のパラメーターの値は1増加します。以下の図1は、レポート中のrank2のタンパク質ファミリー(免疫グロブリンkappa定常領域)にアサインされたいくつかのペプチドの例です。
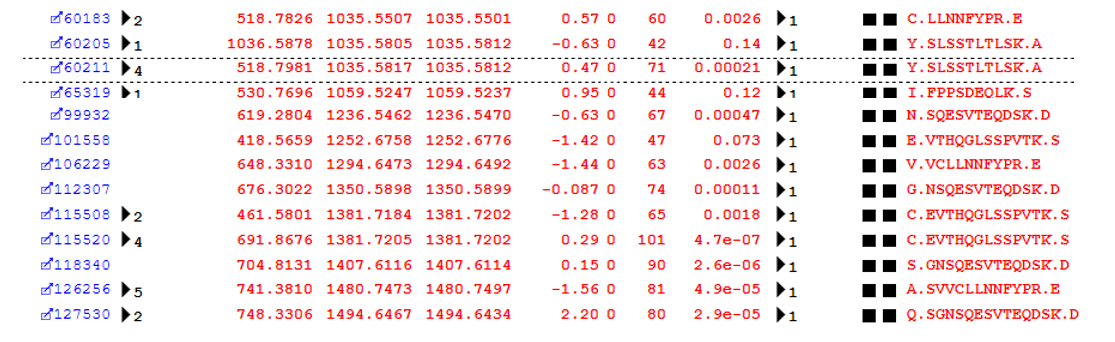 図1: Error Tolerant検索によって同定された半特異的切断ペプチドの例
図1: Error Tolerant検索によって同定された半特異的切断ペプチドの例
Error Tolerant検索はこれらの非特異的切断産物をピックアップするのに非常に効率的な方法です。ライブラリーベースの検索では見落としてしまいがちなペプチドです。
想定外の化学修飾
Error Tolerant検索で同定された修飾のリストには、多数の化学修飾の可能性も含まれています。例えば今回の検索例では、C以外の残基でカルバミドメチル化が同定されたPSMが104個追加されています。このサンプルはヨードアセトアミドでアルキル化されており、過剰なアルキル化はよく知られているため、この表示結果は可能性としては十分あり得ます。アルキル化以外の例では、少し検討が必要なものもあります。Error Tolerant 検索のヘルプページにもう少し詳しい説明がありますが、ここでは1例をピックアップします。
+1の質量、脱アミノ化
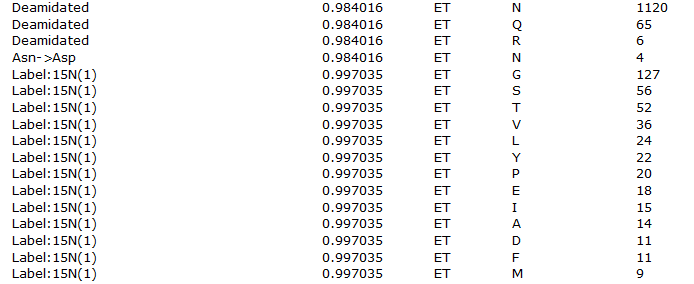
Error Tolerant検索で追加された修飾の表を見ると、下図2に示すように、約+1Daの候補がたくさん挙げられている事がわかります。
この問題については過去のブログ記事「"The plus one dilemma"」でも触れており、その時と同じポイントがここでも当てはまります。脱アミノ化はかなり信憑性の高い修飾ですが、このサンプルは15N標識をしていないため、Label:15N(1)の修飾に関する結果は正しくないでしょう。それよりは他の脱アミノ化や13Cを含むサンプルであった可能性の方が高いでしょう。
翻訳後修飾
リン酸化ペプチドの濃縮など特定の翻訳後修飾の濃縮処理を行った場合は別ですが、Error Tolerant検索はサンプル中のあまり一般的でない翻訳後修飾を同定するためにとても効率的な方法です。今回のデータセットでも、可能性のある翻訳後修飾が多数検出されました:
リン酸化
リン酸化は生物学的に重要な翻訳後修飾のひとつです。Error Torelant検索により、126個のリン酸化ペプチドが同定されました。例としてオステオポンチンのセリン234で同定されたリン酸化をご覧ください(下図3)。リン酸化ペプチドのマッチを示す複数の結果が、ペプチドが溶出しているピーク全般にわたって存在することが示されています。
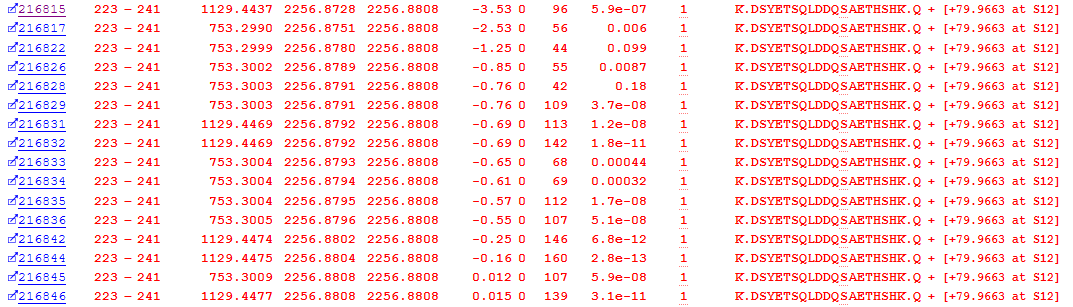
図3:ペプチドDSYETSQLDDQSAETHSHK+リン酸基(セリンに付与)の結果。ペプチドのS12はタンパク質のS234に相当します。
マッチング内容を見ると、実験値と理論値が強力なマッチングをしています。Mascot Delta Scoreの修飾付与部位の解析からも、1位のリン酸化部位の結果に曖昧さがほとんどなく確からしい事を示しています:
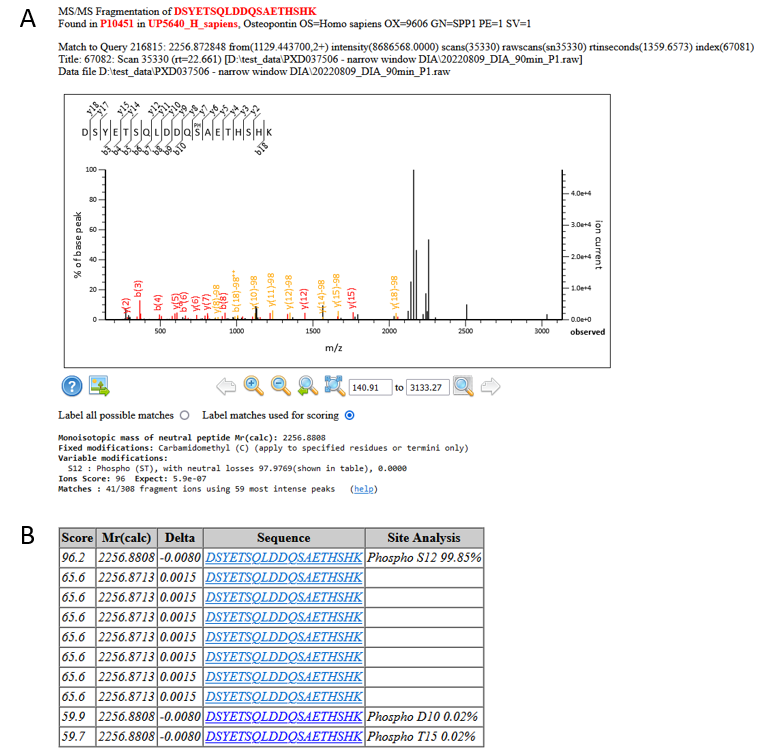 図4: A) DSYETSQLDDQSAETHSHK + S12リン酸化のピークアサイン状況 B) Mascot Delta Scoreによる修飾位置の解析
図4: A) DSYETSQLDDQSAETHSHK + S12リン酸化のピークアサイン状況 B) Mascot Delta Scoreによる修飾位置の解析
Uniprotを見ると、S234はオステオポンチンのリン酸化部位として報告されています。この事からも、結果が妥当である可能性が高いと言えるのではないでしょうか。
グリコシル化
リン酸基の他、生物学的に重要な翻訳後修飾としてグリコシル化が挙げられます。 Mascotでは多くの糖鎖を含む修飾データベースとしてUnimodを使用しており、これによりError Tolerant検索の際、様々な糖鎖を考慮する事ができます。結果画面内で修飾に関する情報をまとめたリストには、383種類の糖ペプチドのPSMsが報告されています。下の図5はアルファ-2-HSグリコプロテインの例で、セリン346に糖鎖Hex(1)HexNAc(1)NeuAc(1) が付いています。
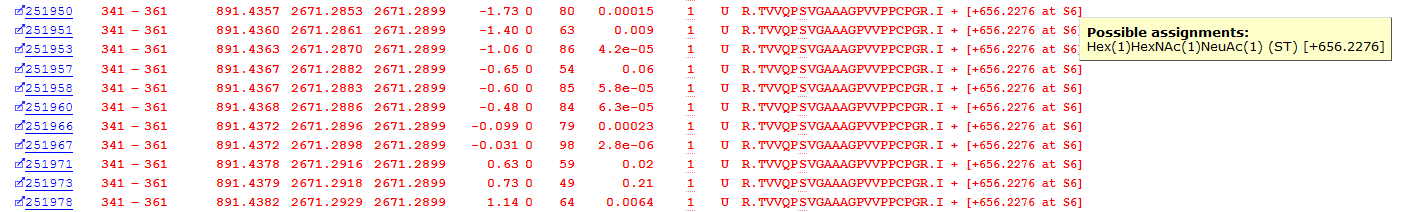 図5:Error Tolerant検索によってリストアップされた、α-2-HS-グリコプロテインの糖ペプチド
図5:Error Tolerant検索によってリストアップされた、α-2-HS-グリコプロテインの糖ペプチド
先程のリン酸化の例と同様、グリコシル化を示す複数の結果が見られます。しかしながら今回の例では、位置の特定が難しい状況です。糖鎖がニュートラルロスしたピークに多くマッチしており、位置特定に必要な情報が不足しているためです。Mascot Delta Scoreを使った特定ができず、糖鎖が付いている位置がS346なのかそれともT341なのか、両者の間に明確な差がない結果となっています。
 図6:A) ペプチドTVVQPSVGAAAGPVVPPCPGR + Hex(1)HexNAc(1)NeuAc(1) のピークアサイン状況B) Mascot Delta Scoreによる修飾位置の解析
図6:A) ペプチドTVVQPSVGAAAGPVVPPCPGR + Hex(1)HexNAc(1)NeuAc(1) のピークアサイン状況B) Mascot Delta Scoreによる修飾位置の解析
UniprotではT341とS346の両方がO-結合型糖鎖付加部位としてリストアップされていました。今回のケースでは判断材料にはなりませんでした。
 図7: Uniprotによるα2-HS-グリコプロテインの糖鎖付加部位情報
図7: Uniprotによるα2-HS-グリコプロテインの糖鎖付加部位情報
GlyConnectをみると、S346のHex(1)HexNAc(1)NeuAc(1)のアサインについては実験的に確認されています。そのためもしどちらかの2択で答えを迫られたらS346を選ぶかもしれません。しかしMascotの今回の検索を根拠にどちらであるかを決定する事はできません。
アミノ酸配列の変異
Error Tolerant検索が対応可能な想定外の変化の例をご紹介してきましたが、最後はタンパク質のアミノ酸配列の変異です。このError Tolerant検索では、1残基置換の可能性のあるPSMsが922個見つかりました。例として、Immunoglobulin heavy constant alpha 2の残基182におけるプロリンからセリンへの置換を下の図8に示します。
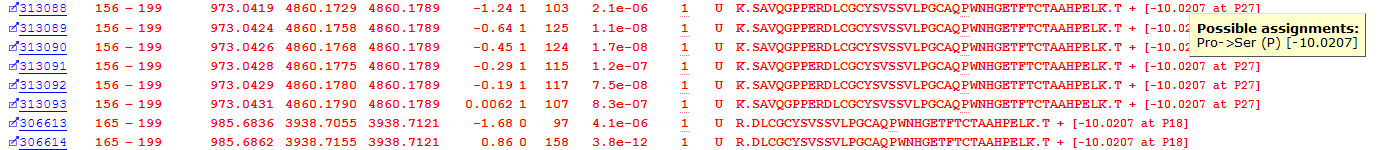 図8: Immunoglobulin heavy constant alpha 2、P182→S182の一残基置換の結果
図8: Immunoglobulin heavy constant alpha 2、P182→S182の一残基置換の結果
同じ残基置換に対して複数の高スコアマッチがあり、Mascot Delta Scoreもその可能性が高いことを示しています。
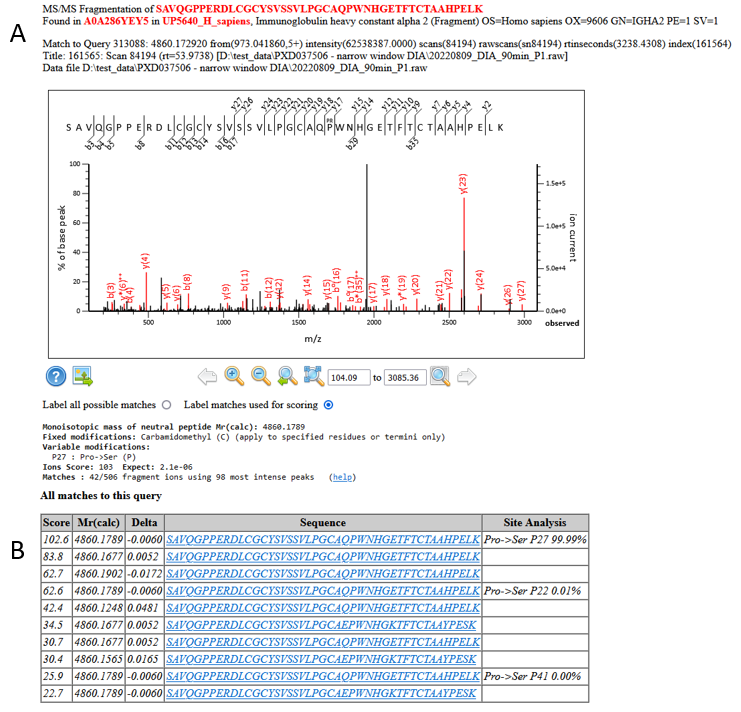 図9: A) P182のPro->Ser残基置換データのピークアサイン状況B) Mascot Delta Scoreによるアミノ酸残基置換位置の解析
図9: A) P182のPro->Ser残基置換データのピークアサイン状況B) Mascot Delta Scoreによるアミノ酸残基置換位置の解析
EBIのProtVarでは、残基182のProlineからSerineへの置換を既知のバリアントとしてリストアップしており、検索結果はこの例と一致します。
結論
ライブラリーが実験的に得られたものであれインシリコで生成されたものであれ、スペクトル中心の検索手法ではカバーが難しい検索空間範囲に対応できる検索方法がError Tolerant検索です。今回狭いウィンドウ幅のDIAデータセットに対するMASCOTのError Tolerant検索を適用する事で、1)アミノ酸配列の変異(1残基置換)、2)想定外の修飾、3)非特異的酵素切断、の3つの可能性を考慮しながら検索可能であることが示唆されました。
Keywords: chimeric spectra, DIA, error tolerant, variable modifications