Mascot DaemonにおけるDDA-PASEFデータを用いたTop-3定量解析
Mascot Daemonには、プリカーサーベースの定量法(LFQ、SILACなど)とMS/MS定量法(iTRAQ、TMT)のどちらにも対応した、定量結果をまとめるサマリーレポート作成機能が含まれています。出力ファイルは、RやPerseusなどの統計パッケージへ渡すことができます。
Mascot Daemon ver.3.0より、「Top-3」法で計算されたタンパク質強度値を出力するオプションを追加しました。マッチしたすべてのペプチドを使用するのでなく、強度値が最も高い上位3つのマッチングペプチドのみをタンパク質の定量計算に使用します。今回は、ベンチマーク用DDA-PASEFデータセットを用いた例についてご紹介します。
DDA-PASEFデータセットによる解析例
Mascot Daemon 3.0のTop-3出力使用例として、PRIDEプロジェクトPXD028735のtimsTOF DDAファイルから1サンプルと3回の繰り返し実験データを入手し再計算しました。
| Species | Log2(Target Ratio) |
|---|---|
| H.sapiens | 0 |
| S.cerevisiae | 1 |
| E.coli | -2 |
ダウンロードした tdf の RAW ファイルの処理と検索は、Mascot Daemon を用いて自動化しました。ピーク抽出処理などのパラメーターについては、timsTOF 向けに準備されたMascot Distiller にあるデフォルト設定を使用しました。timsTOF データを処理する際には、プリカーサーイオンモビリティの情報を使ってを用いて定量中のプリカーサーシグナルをフィルタリングでき、精度の向上および処理の高速化が可能です。詳しくは過去のブログ記事「イオンモビリティフィルタリングによるプリカーサー定量結果の改善 」をご覧ください。
これを Mascot Daemon から利用できるようにするため、Daemonの検索タスクを開始する前に次の設定を行ってください:
- 管理者権限を持つユーザーで Windows にログインします。
- Windows の検索バーに「cmd」と入力します。
- 表示された「コマンドプロンプト」を右クリックし、「管理者として実行」をクリックします。
- MascotDistiller.exe があるディレクトリへ移動します(デフォルト設定では C:\Program Files\Matrix Science\Mascot Distiller です)。
- 次のコマンドを実行します:
MascotDistiller.exe -batch -daemonMGFIonMobilityParam 1
この操作は 最初に一度だけ行えばよく、timsTOF のデータファイルを処理するたびに実行する必要はありません。
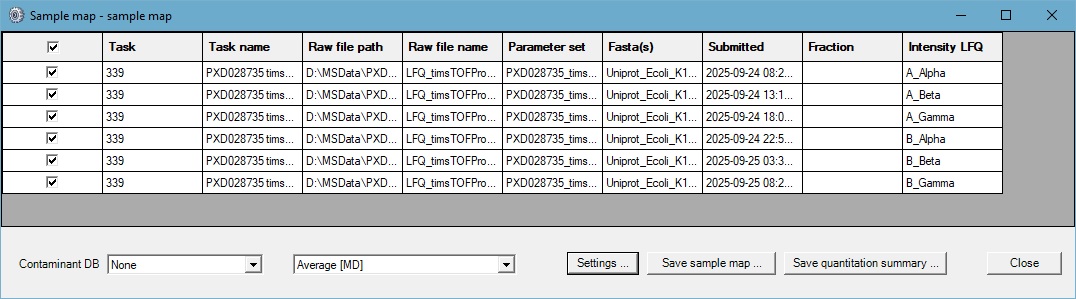
ピーク抽出、検索、そしてラベルフリー定量の “Average” 手法による定量は、Mascot Daemon を用いて自動化しました。PRIDE リポジトリから取得した Sample A および Sample B の最初のバッチについて、Alpha・Beta・Gamma の各繰り返し実験(合計 6 ファイル)の tdf RAW ファイルをデータ処理しました。
その結果、ペプチドFDR 1% の条件で、372,444 件の有意な PSM 、 41,068 個のペプチド配列、および 8,001 個のタンパク質(same-set マッチを除外)が同定されました。
定量サマリーの設定は以下のとおりです:
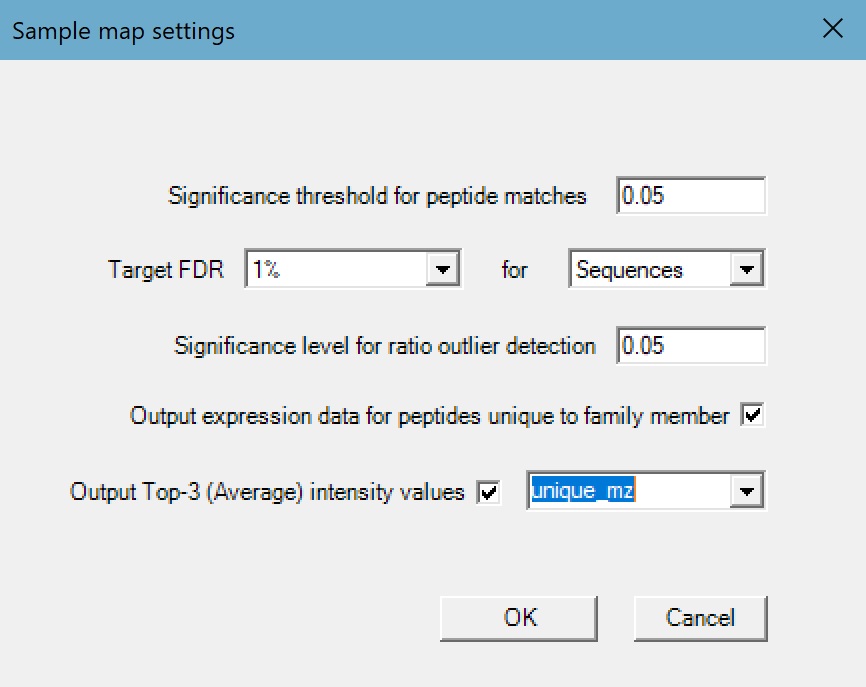
その後、生成されるレポートが ペプチドFDR 1% をターゲットにするよう設定を変更し、Mascot のレポートと一致させました。あわせて、同一タンパク質ファミリー内の特定メンバーに固有なペプチドについて、Top-3 強度値と発現データを出力するようにしました。
これらの設定を有効にすると、生成されるレポートには、割り当てられたすべてのペプチドマッチを用いて算出した各タンパク質の合計強度値とTop-3 強度値、さらにそのタンパク質に固有に割り当てられたペプチドのみを用いて算出した合計強度値とTop-3 強度値が含まれます。
また、Top-3 の計算方法に使用される各ペプチドの定量値ですが、次の 3 つのオプションから選択できます。同じペプチドに対して、電荷や修飾の違いがあったときに別データとみなすかどうかが変わります。
- unique_sequence:同一配列なら、修飾や電荷が違っても合算します。
- unique_mr:配列が同じでかつ修飾も同じなら、電荷が違っても合算します。
- unique_mz:電荷や修飾が異なるものは合算しません。
今回のケースでは、unique_mz を Top-3 計算に用います。これは、他のいずれのオプションよりも同定タンパク質リストをより深く掘り下げることができるためです。
サンプルマップと各オプションの設定が完了したら、「Save quantitation summary …」 ボタンをクリックします。出力されるファイルは タブ区切りのテキスト(TSV) で、Excel で取り込む事ができます。
下表は、タンパク質の合計強度および Top-3 強度について、そのタンパク質に固有に割り当てられたペプチドのみを用いて算出した Sample A / Sample B の中央値 log2 比です。
| Species | Median Log2(Total Intensity A / Total Intensity B) | Median Absolute Deviation | N | Median Log2(Top 3 Intensity A / Top 3 Intensity B) | Median Absolute Deviation | N |
|---|---|---|---|---|---|---|
| H.sapiens | -0.167 | 0.291 | 4661 | -0.117 | 0.191 | 2688 |
| S.cerevisiae | 0.943 | 0.441 | 1561 | 0.725 | 0.238 | 606 |
| E.coli | -2.475 | 0.702 | 208 | -1.819 | 0.362 | 66 |
Top-3 強度は、ヒトおよび E.coli のタンパク質については、目標比により近い中央値比を与えますが、酵母のタンパク質ではやや異なる数値となっています。酵母タンパク質の結果が他の 2 種に比べて良好でない理由については、今後さらに検討します。
しかし、いずれの場合でも中央値平均偏差(median average deviation)は低く、計算された比のばらつきは小さくなっています。これは一部、Top-3 法では最も強度の高い 3 本のペプチド配列の強度のみを用いて計算するため、外れ値的なペプチド強度の影響を受けにくいことによります。
このアプローチの主な欠点は、有意なペプチド配列マッチが3つ未満のタンパク質についてはタンパク質強度値が得られない点です。その結果、レポート下位に並ぶタンパク質では強度が計算される件数が少なくなります――これは、上表で各生物種の中央値を算出するのに用いられたタンパク質数が少ないことからも明らかです。
Keywords: ddaPASEF, Mascot Daemon, Mascot Distiller, quantitation, quantitation summary, timsTOF