LC-MS/MSデータのMascotワークフロー
質量分析ベースのプロテオミクスのデータ解析は複雑であるため、現在ではほとんどソフトウェアがその役割を担っています。rawファイルの処理、データベース検索によるペプチドの同定、タンパク質の推定、タンパク質の定量といった計算には、膨大な数のパラメーターが存在する事は言うに及ばず、多くの計算過程・組み合わせが存在します。各プロセスでの最適な解析方法同様、ソフトウェアも進化し続けています。質量分析プロテオミクスの初心者も熟練者も、一歩引いて定期的に基本を学ぶことが重要です。
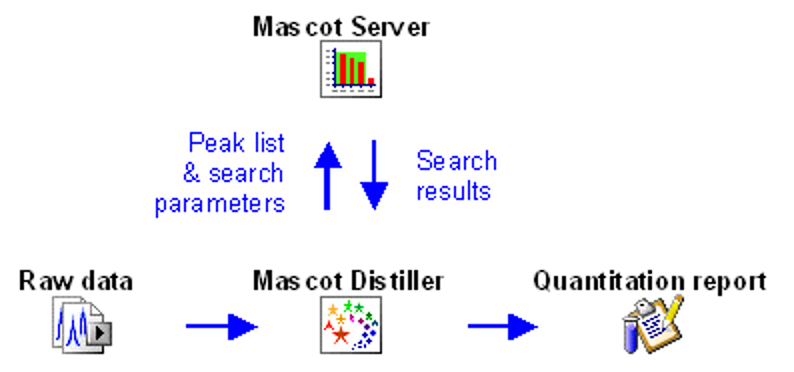
ピーク抽出
データ解析の出発点は、質量分析計の測定から得られた「raw」ファイルです。rawファイルには、survey(MS)スキャンと、それに続く1つまたは複数のMS/MSスキャンが含まれています。ボトムアッププロテオミクスの目標は、サンプル中に存在するすべてのペプチドを同定することです。ほとんどのソフトウェアでは、MS/MSスペクトルをタンパク質配列データベースまたはスペクトルライブラリーに対して検索することで実行されます。
Mascot Serverはrawデータを入力データとして受け付けて中身を読み取る事ができません。最初の解析はrawファイルをピークリストへ変換する事です。これは、検索に無関係なノイズ、モノイアソトピック以外の同位体ピーク、解析に利用しない電荷のピークを除去します。そうすることで、データ解析下流に行く前にファイルサイズを縮小しMS/MSスペクトル内の重要なメタデータをエンコードします。装置のデータシステム側でピーク抽出を行ってくれる場合もありますし、それ以外の解析ツールを使用することもできます。弊社製品Mascot Distillerは、どのような装置のrawデータでも動作するよう設計されています。
チュートリアル”How to configure Mascot Distiller peak picking”では、MSおよびMS/MSのピーク抽出のオプションについて網羅的に説明しています。Mascot Distillerには全ての質量分析装置向けのデフォルトパラメータセットが同梱されています。過去のブログ記事「Peak picking Thermo .RAW data with Mascot Distiller」(英語版、日本語版)では、Thermofisher 社装置向けの2つのパラメータセットを適用した場合の結果の違いを検証しています。Distillerを初めてお使いになる場合、RAWファイルをお送りいただいて、弊社側で適切な処理オプションファイルをご案内する事も可能です。詳細は support-jp@matrixscience.com までお問い合わせください。
サンプルがDDA(Data Dependent Acquisition) モードで測定された場合でも、MS/MSスペクトルの一部で、複数のペプチド由来のフラグメントが混ざって測定されることがあります。一方DIA (Data Independent Acquisition) モードで測定された場合は、そのほとんどのMS/MSに複数のペプチド由来のピークが含まれるでしょう。Mascot Distillerでは、複数のペプチドが混ざった”キメラ”DDAデータを処理する事が可能(英語版、日本語版)ですし、DIAデータにおいても分離ウィンドウが8m/z以下のナローウィンドウのデータ解析(英語版、日本語版)であれば対応しています。どちらの場合も、Distillerはプリカーサーに関する情報をMascot検索に適したフォーマットであるmgfファイルとしてまとめ直します。
データベース検索
チュートリアル「Searching uninterpreted MS/MS data」では、Mascotデータベース検索パラメーターと配列データベースの選択方法について説明しています。パラメーターの検証に必要な事は何か?BSA 消化物のような標準サンプルを測定・検索実行し、その結果を元に最適な検索パラメーターを確認・設定することです。未知のサンプルに対して検索パラメーターを設定するのはより難しい問題ですが、過去のブログ記事「Back to Basics: Optimize your search parameters」で説明されているように、Mascotにはそのためのツールがすべて含まれています。
検索エンジンは、各ペプチドにマッチするデータベースエントリーが、観測されたMS/MSスペクトルとどの程度一致するかを反映するスコアを与えます。Mascot はピークと理論値のマッチング内容が偶然に起こる確率(p)を計算します。スコアは単純に -10log10(p)となります。過去のブログ記事「Back to basics 5: Peptide-spectrum match statistics」では、その詳細と意味について説明しています。
確率的スコアリングは「高品質な検索結果」に対する必要条件といえますが、十分条件ではありません。Mascotを検索する時、パラメーター「Decoy」には常にチェックする事をお勧めします。有効にするとMascotは、ピークリストを通常のターゲットデータベースと、配列が逆向きまたはランダム化されたデコイデータベースの両方に対して検索します。デコイデータベースのマッチ数は、ターゲットデータベースの結果における偽陽性の数の優れた推定値として扱われ、”FalseDiscovery Rate”が報告されます。
また、検索エンジンMascot Serverに統合されているPercolatorを使用して、ターゲット、デコイの両結果を元にした再スコアリングすることもお勧めします。Percolatorは半教師付き機械学習を使って、ターゲットマッチとデコイマッチの最適な分離方式並びに分離点を見つけます。FDR同様デコイマッチがターゲットマッチの偽陽性の数として遜色ないモデルとみなすことができるかが重要です。みなすことが出来る場合、この再スコアリングにより、感度(ここでは、統計的に有意とみなされた基準を超える正しいターゲットマッチの数、と置き換えて考えてください)と特異度(ターゲットマッチの中で統計的基準を超えていない、不正解の数)の両方を向上させる事ができます。特に内因性ペプチドのような "解析が難しい "データセットでは有益です。ブログ記事「Identify more HLA peptides」(英語版、日本語版)に利用例がございますので併せてご覧ください。
タンパク質の推定
データベース検索はペプチド配列を同定します。プロテオミクスで使用されるすべての検索エンジンはタンパク質を同定するために、タンパク質の推定と呼ばれる計算過程を経る必要があります。デフォルトでは、Mascotは、ヒットしたタンパク質にユニーク(他のタンパク質配列と共有されていない)で、統計的に有意なスコアを持つペプチドが少なくとも1つマッチするタンパク質のみが結果に表示されます。ユニークなペプチドを持たないタンパク質(subsetタンパク質)は非表示になります。Mascotは、2つ以上のタンパク質が全く同じ同定ペプチドを共有するsame setタンパク質に関する情報も表示します。マススペクトル内にはsame setのうちどちらなのかを判断できる情報がありません。タンパク質のアクセッション(ID情報に相当)をデータベース内で事前に英数字順にソートしておくことで「リード」タンパク質が選択され、表示されます。
Mascotはヒットしたタンパク質に対して常にタンパク質スコアを表示しますが、これは直接的には確率的な意味合いがないスコアです。ブログ記事「Common myths about protein scores」では、文献に繰り返し出てくるいくつかの誤解について言及しています。サンプルがそれほど複雑でない場合、MascotのReport Builderを使用して、同定されたタンパク質のリストを作成する事は非常に簡単です。一つ付け加えるとするなら、多くのジャーナルで要求されるProtein FDRの値もチェックし、少なくとも数字を記録しておく必要があります。
検索するデータベースが大きければ大きいほど、samesetタンパク質やsubsetタンパク質が存在する可能性が高くなります。サンプルが単一の生物種から得られたものであれば、検索後の結果画面で”preferred taxonomy”の設定を適用する事により、適切な生物種からのタンパク質の表示を優先させる事ができます。複雑なサンプルの場合、どのsame setタンパク質がサンプルに本当に含まれているかを判断するには、実験に対して追加の検証が必須となります。関連するブログ記事(英語版>/a>、日本語版)も併せてご覧ください。
定量
ほとんどの実験では、タンパク質を同定するだけでなく、サンプル間にわたる相対量を定量解析する事も求めているでしょう。Mascotにおける定量解析の哲学は、まず同定し、その結果をベースに定量することに重きを置いています。すなわちまずデータベース検索でマッチした同定ペプチドのリストを作成し、定量解析を行うのに遜色ない基準を満たすペプチドを対象に計算を行います。そしてペプチドの定量情報を元に、ペプチドがアサインされたタンパク質の定量値も計算します。
現在最もポピュラーなプロトコルは、ラベルフリー定量(LFQ)とiTRAQ/TMTラベリングです。iTRAQ/TMTラベリングは、MS/MSスペクトル中にレポーターイオンを生成し、その相対強度を各サンプル中のペプチド強度として使用します。プロトコルに関係なく、タンパク質はペプチド強度または強度比に基づいて定量されます。「Back to basics 3: Quantitation statistics」では、その背景にある古き良き統計的手法について説明しています。
Mascot Distillerは他の定量計算プロトコルもサポートしています。Distillerによる定量計算について興味がある方は、弊社のオンライン資料「Quantitation module」をご覧ください。また、最適なアプローチについてご質問があれば、メールにてお問い合わせください。
計算で得られた数値の表を得る事が解析のゴールとはなりません。ブログ記事「Reporting quantitation datasets with Mascot Distiller 2.8」(英語版、日本語版)では、MASCOT Distillerに組み込まれたレポート機能で何ができるかをご紹介しています。また、RやPerseusでさらに処理するためにCSV形式でデータをエクスポートしたり、Mascot Daemonで複数の解析から得られた発現データを集計したりすることもできます。Distillerのファイル(.XML, .rov)は、Scaffold DDAのようなサードパーティのツールでインポートして更なる解析に繋げる事もできます。
Keywords: Mascot Distiller, peak picking, protein inference, quantitation, scoring, statistics, tutorial